Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
En collaboration avec des chercheurs de l'Institut Weizmann en Israël, une équipe de l'Institut de génétique humaine (CNRS) vient de révéler pour la première fois l'architecture tridimensionnelle fine des chromosomes (1): Giacomo Cavalli et ses collègues ont réussi à cartographier à haute résolution (2) les différents contacts existant à l'intérieur et entre les chromosomes. Un défi relevé grâce à une nouvelle approche d'analyse génétique à très haut débit améliorée par l'équipe montpelliéraine. Ces travaux de recherche majeurs devraient permettre de mieux comprendre l'impact de l'organisation tridimensionnelle des chromosomes sur l'expression du génome (3) et sur la survenue de maladie comme les cancers. Ils ont été publiés dans la version en ligne de la revue Cell du 19 Janvier 2012.
Les chercheurs s'en doutaient depuis quelques années déjà: un individu est plus que la somme de ses gènes ; l'architecture de ses chromosomes serait aussi un facteur clé pour le bon fonctionnement de ses cellules. Mais jusqu'ici, une réelle compréhension des principes qui organisent le repliement du génome dans le noyau échappait aux analyses. Et depuis 4 ans, une vingtaine d'équipes dans le monde tentaient de mettre au point une technique d'analyse pour étudier ce repliement.
La méthode de l'équipe CNRS dérive d'une autre développée par une équipe américaine en 2009. Une technique baptisée "capteur de la conformation des chromosomes" (Hi-C), qui permet de décrire les contacts que chaque région du génome établit avec les autres. Comme la version américaine, la méthode montpelliéraine consiste en trois grands étapes: figer les contacts dans ou entre les chromosomes avec le composé formaldéhyde (4) ; lier les bouts des deux gènes en contact avec la molécule ADN ligase (5), d'origine bactérienne ; et déterminer la séquence de ces deux gènes. Mais Giacomo Cavalli et ses collègues y ont apporté d'importantes améliorations rendant possible l'étude de fragments d'ADN beaucoup plus petits (de quelques centaines de paires de bases, contre des fragments de 5-10 kilobases avant). Ce qui permet de cartographier ces contacts à haute résolution.
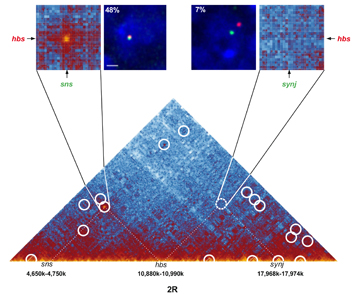
Exemple de carte d'interaction tridimensionnelle pour une région génomique d'une taille de 15 Millions de paires de bases (environ un quart d'un chromosome, ici le chromosome 2R). Les cercles entourent des régions très distantes à l'échelle linéaire mais qui établissent des très forts contacts dans les cellules. En haut à gauche de la figure, un encart montre un de ces contacts cartographié à haute résolution et impliquant deux gènes (hbs et sns) réprimés dans les mêmes cellules. Juste à sa droite, un autre encart montre une image de microscopie où les deux gènes en question ont été colorés en vert et en rouge et colocalisent à l'intérieur dans 48% des cellules embryonnaires.
La même analyse est montrée dans les encarts à droite pour deux autres gènes (hbs et synj) caractérisés par des contacts beaucoup plus faibles en microscopie ainsi qu'avec la méthode de HiC, malgré le fait qu'ils soient à la même distance linéaire le long du chromosome. Ces cartes, dont on dispose désormais pour l'ensemble des gènes, permettront d'étudier comment l'environnement tridimensionnel influence l'activité de chacun des gènes dans des cellules normales ou pathologiques.
© Giacomo Cavalli (CNRS)
Avec cette technique, les chercheurs montpelliérains et leurs collègues israéliens ont analysé le génome de la mouche du vinaigre ou drosophile. Un animal avec un génome quinze fois plus petit que le nôtre. Résultat: l'équipe a pu y identifier... 118 millions de zones de contact ! L'analyse de ces zones leur a permis ensuite d'établir des cartes d'interaction très détaillées.
Ces cartes montrent que les chromosomes sont organisés en domaines contenant un ou plusieurs gènes. Séparés par des régions frontières, ces domaines correspondent soit à des zones activement exprimées (6), soit à des régions inactives.
Grâce aux compétences en mathématiques de l'équipe israélienne de Amos Tanay, les deux équipes ont pu ensuite développer un modèle informatique capable de prédire assez fidèlement le repliement réel des chromosomes. Très puissant, cet outil a permis de décrypter les principes fondamentaux qui régissent l'organisation tridimensionnelle du génome. Notamment, il est apparu que les domaines inactifs ont tendance à former des contacts avec d'autres domaines inactifs souvent proches sur le même chromosome, alors que les domaines actifs "contactent" d'autres domaines actifs lointains ou proches et éventuellement sur d'autres chromosomes.
Cruciaux, ces travaux devraient permettre de mieux comprendre les différences de repliement chromosomiques selon les types de cellules (cellules de foie, de muscle, nerveuse, etc.) et l'importance de ce repliement dans la survenue de cancers.
Notes:
(1) Supports physiques de l'information génétique, constitué d'ADN. Le noyau de nos cellules en renferme 23 paires.
(2) Quelques centaines de paires de bases.
(3) L'ensemble du matériel génétique d'un individu.
(4) Composé chimique dérivé du formol qui a la faculté d'arrêter toute activité cellulaire et de former des ponts covalents entre protéines et ADN.
(5) Enzyme qui répare les brins brisés d'ADN.
(6) Zones qui donnent lieu à la production de protéines.
Référence:
Three-dimensional folding and functional organization principles of the Drosophila genome. Giacomo Cavalli et co. Cell online du 19 janvier 2012
Populaires