Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
Illustration: CNRS
Pour déterminer comment sont repliés les deux mètres d'ADN qui se trouvent dans nos cellules, les biologistes moléculaires ont mis au point dans les années 2000 la technique de "Capture de Conformation des Chromosomes". Cette méthode mise en œuvre dans des cellules vivantes fournit la localisation de toutes les zones du brin d'ADN se trouvant en contact l'une avec l'autre. Les techniques développées jusqu'à présent pour reconstituer la structure tridimensionnelle des chromosomes à partir de ces informations reposaient sur des méthodes d'optimisation progressive qui ne pouvaient fonctionner qu'avec un jeu partiel de données et requerraient un temps de calcul prohibitif.
Des physiciens théoriciens du Laboratoire de physique théorique de la matière condensée - LPTMC (CNRS/UPMC) viennent de mettre au point une méthode de calcul directe, qui s'accommode de données incomplètes ou bruitées et qui est bien plus rapide que les méthodes actuelles. La structure tridimensionnelle obtenue peut ensuite être croisée avec d'autres jeux de données génomiques, par exemple la position de certaines protéines le long du génome pour visualiser les structures formées en trois dimensions par ces protéines dans le noyau. Ce travail est publié dans la revue Nature Methods.
Pour déterminer les zones de contact, les biologistes fixent celles-ci chimiquement puis après avoir découpé l'ADN ainsi fixé, ils déterminent les régions du brin d'ADN qui sont concernées par ces contacts. La méthode mise au point par les physiciens statisticiens du LPTMC s'effectue en plusieurs temps. Dans un premier temps, ils utilisent ces données pour engendrer un graphe dans lequel deux sites seront reliés par un lien s'ils sont en contact. De ce graphe, ils définissent comme distance topologique entre deux sites quelconques le nombre de liens du graphe par lequel passe le plus court chemin reliant ces deux sites et construisent une matrice auxiliaire à partir de ces distances.
C'est la détermination des trois premiers vecteurs propres de cette matrice qui leur fournit alors les coordonnées tridimensionnelles des points de contact. La comparaison des résultats de cette approche avec d'une part des simulations numériques, et d'autre part le résultat de mesures sur des polymères ou de l'ADN, a permis aux chercheurs de valider le fait que cette méthode permet effectivement d'utiliser une information purement topologique (le nombre de liens dans un graphe) pour obtenir une configuration géométrique qui ne présente aucune déformation. Par son principe, c'est-à-dire la recherche de vecteurs propres, cette méthode est directe et ne nécessite qu'une seule étape, contrairement aux méthodes d'optimisations utilisées jusqu'à présent. De plus, comme seulement trois vecteurs propres sont requis, cette méthode s'accommode très bien de données lacunaires et/ou bruitées.
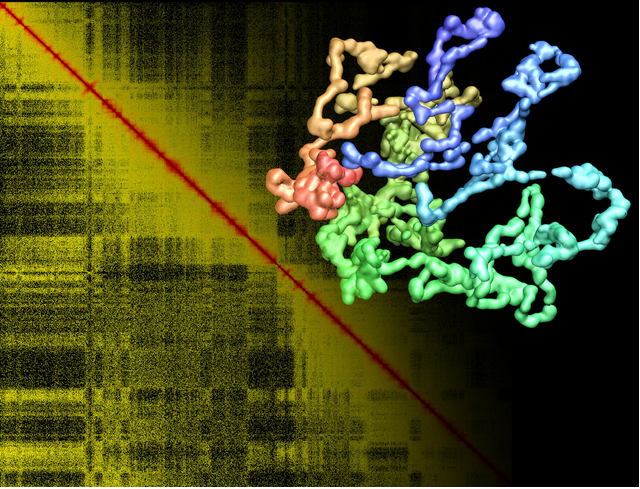
le fond montre une carte de contacts chromosomiques humains après traitement et normalisation des données expérimentales obtenues in vivo: plus un pixel est de couleur rouge, plus la fréquence de contact entre les deux sites génomiques associés (en abscisse et en ordonnée) est élevée. L'algorithme proposé par Lesne et al. permet de reconstruire la structure 3D correspondante, ici le chromosome 1 où le gradient de couleur indique la position le long du génome.Illustration: CNRS/INP
Pour plus d'information:
- Voir-les-chromosomes-en-3-dimensions
- 3D genome reconstruction from chromosomal contacts
Populaires