Dieser bedeutende wissenschaftliche Durchbruch, das Ergebnis einer internationalen Zusammenarbeit im Rahmen des 4D Nucleome Project, enthüllt die dreidimensionale und dynamische Organisation des menschlichen Genoms. Durch die Untersuchung von embryonalen Stammzellen und Fibroblasten erfassten die Forscher die Prinzipien, die regieren, wie sich Chromosomen im begrenzten Raum des Zellkerns falten und interagieren und so direkt die Aktivität der Gene beeinflussen.

Die innige Architektur des Zellkerns
Im Gegensatz zum linearen Bild, das von der Sequenzierung stammt, nimmt die DNA eine extrem organisierte räumliche Konformation an. Sie bildet Schleifen, Domänen und Kompartimente, deren Geometrie den Zugang zu den molekularen Maschinen bestimmt, die für das Lesen der genetischen Information zuständig sind. Diese 3D-Struktur ist nicht zufällig; sie legt fest, welche Gene aktiviert oder unterdrückt werden und formt so die Identität und Funktion jeder Zelle.
Um diese Landschaft zu kartieren, fusionierten die Wissenschaftler Daten mehrerer modernster genomischer Technologien. Dieser integrative Ansatz ermöglichte es, die Grenzen jeder einzelnen Methode zu überwinden. Das Ergebnis ist eine Reihe hochauflösender Modelle, die die architektonische Variabilität von Zelle zu Zelle erfassen und so eine nuanciertere und realistischere Sicht auf das intranukleare Leben bieten.
Die Arbeit, veröffentlicht in Nature, identifizierte mehr als 140.000 Interaktionen in Form von Chromatin-Schleifen pro Zelltyp. Diese Schleifen bringen DNA-Sequenzen, die weit voneinander entfernt sind, physisch näher zusammen, wie genetische Schalter mit den Genen, die sie kontrollieren. Die präzise Karte dieser molekularen Ankerpunkte ist ein Schlüssel zum Verständnis der grundlegenden Mechanismen der Genregulation.
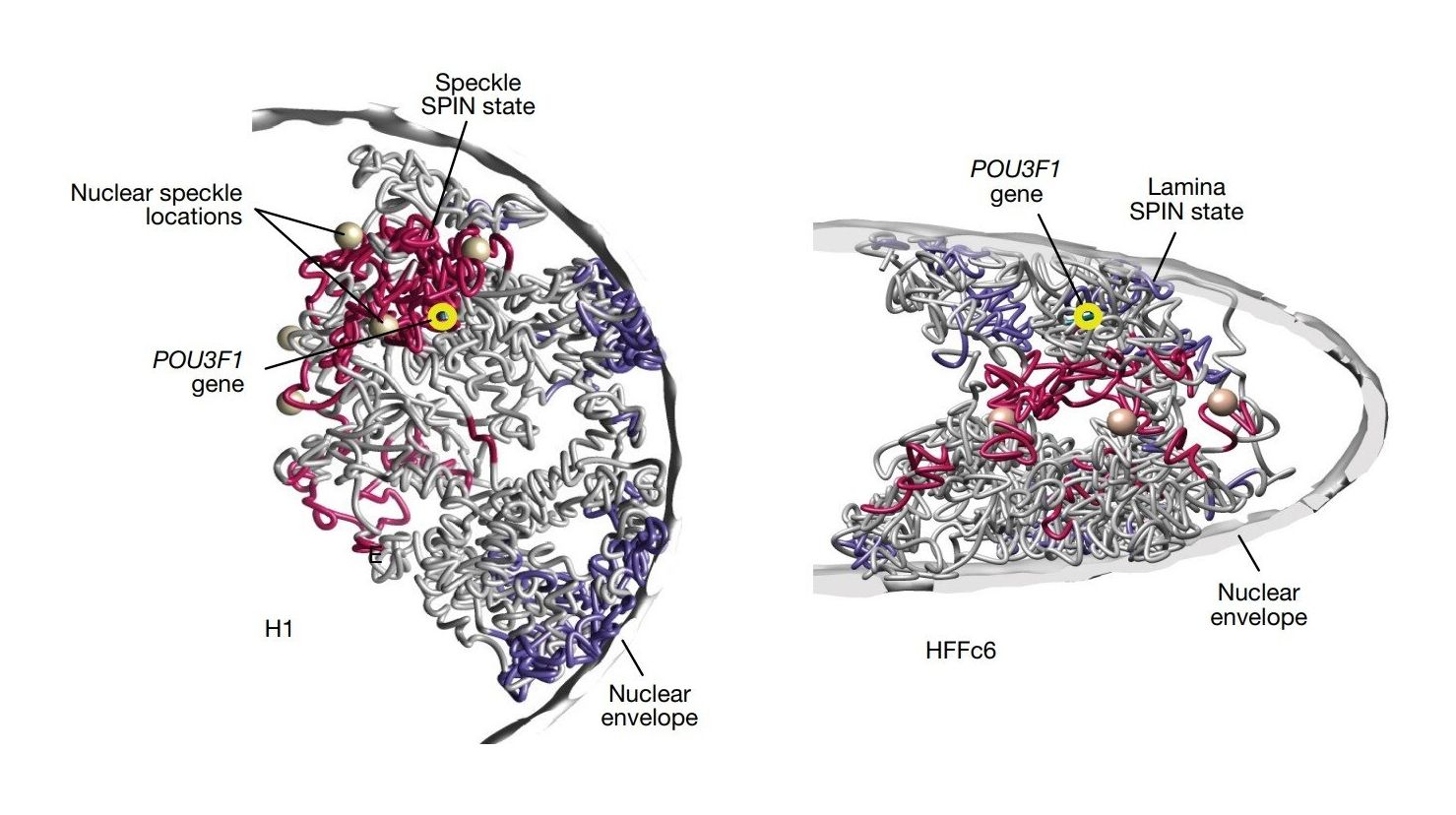
Repräsentative 3D-Einzellstrukturen von Chromosom 1 in H1-Zellen (links) und HFFc6-Zellen (rechts).
Quelle: Nature (2025)
Die Auswirkungen von Mutationen durch die Form vorhersagen
Einer der bedeutendsten Beiträge dieser Studie liegt in der Entwicklung computergestützter Werkzeuge, die vorhersagen können, wie sich eine DNA-Sequenz falten wird. Dieser Fortschritt eröffnet die Perspektive, die Auswirkung genetischer Variationen auf die Genomstruktur vorherzusagen, ohne auf Laborexperimente zurückgreifen zu müssen. Dies ist wesentlich, um Mutationen in nicht-kodierenden Regionen zu interpretieren, die oft an Krankheiten beteiligt sind.
Tatsächlich verändern viele mit Krankheiten wie Krebs oder Entwicklungsstörungen assoziierte Variationen nicht direkt ein Gen, sondern stören wahrscheinlich seine strukturelle 3D-Umgebung. Die neue Kartierung bietet einen Rahmen, um zu identifizieren, welche Gene von diesen fernwirkenden Veränderungen betroffen sein könnten, und verbindet so kryptische Mutationen mit ihren biologischen Folgen.
Die Forscher haben auch einen rigorosen methodischen Leitfaden etabliert, indem sie die Leistungsfähigkeit der verschiedenen Techniken zur Genomkartierung verglichen. Dieses Referenzwerk ermöglicht es der wissenschaftlichen Gemeinschaft, die am besten geeigneten Werkzeuge für die Untersuchung eines spezifischen Aspekts der nuklearen Organisation zu wählen, sei es der Schleifen, der Chromosomendomänen oder der Positionierung im Kern.
Das Ziel dieser Arbeiten ist translational. Nachdem sie strukturelle Anomalien des Genoms bei Leukämien oder Hirntumoren beobachtet haben, zielt das Team nun darauf ab, zu erforschen, wie diese Architektur pharmakologisch angegriffen werden kann, zum Beispiel mit epigenetischen Inhibitoren. Das Verständnis der Form des Genoms könnte so zu neuen diagnostischen und therapeutischen Strategien führen.