
Die entscheidende Rolle der genetischen Rekombination
Die sexuelle Fortpflanzung ermöglicht eine genetische Vermischung der Eltern durch einen Prozess namens Meiose. Während dieser Zellteilung tauschen die Chromosomen DNA-Segmente aus. Die Rekombination zwischen elterlichen Chromosomen und ihre Verteilung in den Keimzellen hat langfristige Auswirkungen auf die Evolution der Genome. Insbesondere erhöht die Rekombination die Effizienz der Selektion und erleichtert die Anpassung.
Allerdings sind die Orte, an denen diese Austausche stattfinden, nicht zufällig gewählt. Zwei Hauptmodelle oder Muster der Verteilung wurden identifiziert. Das erste, das bei vielen Arten (Pflanzen, zahlreiche Metazoen) beobachtet wird, basiert auf zugänglichen Bereichen des Chromatins, die größtenteils regulatorische Regionen der Genexpression sind. Das zweite wird durch ein spezielles Protein namens PRDM9 bestimmt, das sich an spezifische DNA-Sequenzen bindet, um dort die Rekombination auszulösen.
PRDM9: ein Gen mit paradoxer Rolle
Dieser PRDM9-abhängige Mechanismus wurde bisher nur bei bestimmten Säugetieren beschrieben und weist paradoxe Eigenschaften auf. Obwohl er die Rekombinationsstellen bestimmt, unterliegen diese einer Erosion über die Generationen hinweg. Dieses Phänomen führt zu einem endlosen evolutionären Wettlauf, bekannt als "Red-Queen-Modell", der die Selektion neuer PRDM9-Allele beinhaltet, die neue Stellen im Genom erkennen, die ihrerseits wieder erodieren.
Dieser durch PRDM9 verursachte genetische Konflikt ist umso überraschender, da viele Arten ohne dieses Gen korrekt rekombinieren können (erstes Muster). Darüber hinaus zeigt die phylogenetische Studie von PRDM9, dass dieses Gen im Laufe der Evolution mehrmals unabhängig voneinander verloren ging, möglicherweise im Zusammenhang mit der Erosion der Stellen.
All dies wirft die Frage nach seiner tatsächlichen Rolle im Leben auf. Daher war es zunächst wichtig zu bestimmen, ob die Funktion von PRDM9 auf Säugetiere beschränkt ist.
Eine überraschende Entdeckung bei Salmoniden
Um diese Frage zu beantworten, analysierten die Wissenschaftler in einem in der Zeitschrift PLOS Biology veröffentlichten Artikel die Phylogenie von PRDM9 bei Wirbeltieren und identifizierten, dass ein Homolog dieses Gens bei Salmoniden vorhanden ist, während es bei nahe verwandten Fischen fehlt. Sie entwickelten eine Reihe molekularer und populationsgenetischer Ansätze, die es ihnen ermöglichten, festzustellen, dass PRDM9 tatsächlich eine Rolle bei der Bestimmung der Rekombinationsstellen spielt.
Es weist eine hohe allelische Vielfalt in seiner DNA-Bindungsdomäne auf. Darüber hinaus identifizierten sie molekulare Marker, die charakteristisch für die Aktivität von PRDM9 sind, wie chemische Modifikationen der Histone, der mit DNA assoziierten Proteine.
Durch die Untersuchung verschiedener Arten und Populationen von Lachsen (Atlantik, Pazifik, Ostsee) konnten sie eine Divergenz der Rekombinationsstellen zwischen den am weitesten entfernten Populationen nachweisen, was genau zu erwarten ist, wenn PRDM9 aktiv ist. Ebenfalls in Übereinstimmung mit dieser entscheidenden Rolle von PRDM9 konnten die Wissenschaftler beim Lachs DNA-Sequenzen identifizieren, die spezifisch an Rekombinationsstellen angereichert sind, und eine Erosion dieser Sequenzen im Laufe der Evolution feststellen.
Diese Beobachtungen bestätigen, dass das PRDM9-Gen, weit davon entfernt, auf Säugetiere beschränkt zu sein, bereits vor mehreren hundert Millionen Jahren aktiv war.
Perspektiven: die Ursprünge von PRDM9 ergründen
Somit ist die Funktion von PRDM9 für die Lokalisierung von Rekombinationsstellen nicht auf Säugetiere beschränkt und entstand viel früher als erwartet, mit den damit verbundenen genetischen Konflikten, die sich über mehrere hundert Millionen Jahre hinweg fortsetzen. Es wird wichtig sein, weiter in der Evolution zurückzugehen, um das Auftreten von PRDM9 zu identifizieren und die selektiven Kräfte zu verstehen, die zu seiner Erhaltung beitragen.
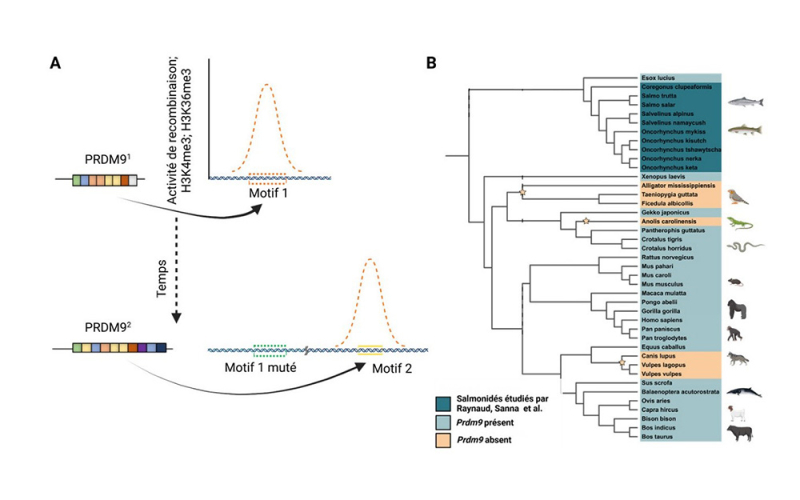
A. Dynamik und Evolution der Rekombinationsstellen. PRDM9 bindet durch seine Zinkfingerdomäne (farbige Quadrate) an spezifische DNA-Sequenzen (Motiv 1). An diesen Stellen gibt es eine Anreicherung von H3K4me3 und H3K36me3 sowie eine hohe Rekombinationsaktivität. Die Rekombinationsaktivität führt zur Akkumulation von Mutationen an diesen Stellen, die nicht mehr vom ursprünglichen PRDM9-Allel (hier PRDM91) erkannt werden. Neue PRDM9-Allele entstehen (hier PRDM92), die unterschiedliche Motive erkennen. Die Rekombinationsstellen werden dann durch die Stellen ersetzt, die von diesem neuen Allel erkannt werden.
B. PRDM9 ist bei Salmoniden und einigen Säugetieren aktiv. Es ist vorhanden, aber seine Aktivität wurde bei anderen Arten (z.B. Gekko) nicht getestet. PRDM9 fehlt in vielen anderen Phyla. Hier sind drei Verlustereignisse während der Evolution durch einen Stern gekennzeichnet.
@ Erstellt in BioRender.
De massy, B. (2025)