
Imagen de ilustración Pixabay
La estabilidad del material genético es esencial para el buen funcionamiento de cada una de nuestras células. Sin embargo, pueden ocurrir accidentes, por ejemplo, bajo la influencia de radiaciones o errores durante la replicación del ADN. Para reparar estas roturas existen mecanismos extremadamente conservados en todas las especies que se encargan de realizar una reparación fiel.
La recombinación homóloga es una vía de reparación de alta fidelidad de las roturas que utiliza una molécula de ADN intacto de secuencia idéntica como plantilla. Esta secuencia puede provenir de la cromátida hermana, del cromosoma homólogo más distante, o de otras secuencias repetidas en el genoma. Por tanto, esta copia homóloga intacta debe ser identificada en la inmensidad del genoma y del espacio nuclear. ¿Cómo se lleva a cabo esta búsqueda de una aguja homóloga en el pajar del genoma?
Para responder a esta pregunta, científicos del CNRS desarrollaron una técnica de genómica de alto rendimiento llamada ssHi-C en la levadura Saccharomyces cerevisiae, un modelo para el estudio de los mecanismos fundamentales del mantenimiento del ADN. Esta técnica les permitió cartografiar, a escala del genoma, los contactos hechos por la rotura. Estos datos permitieron revelar que la búsqueda de homología procedía en dos grandes fases:
- una primera fase en la que la estructuración del genoma por las cohesinas restringe la búsqueda localmente, siendo las cohesinas proteínas que aseguran la cohesión de las cromátidas hermanas al atrapar el ADN en su interior.
- una segunda fase durante la cual la búsqueda se emancipa de estas restricciones y explora sitios genómicos distantes. Esta búsqueda de largo alcance es posible gracias a la producción de largas cadenas monocatenarias de ADN a ambos lados de la rotura, y su rigidificación mediante la formación de un filamento de la recombinasa Rad51. Esta proteína, que cataliza los eventos de recombinación, actúa en este caso como una caña de pescar y lanza su línea, formada por largas cadenas de ADN monocatenario, lejos en el núcleo. Los dos extremos de la rotura buscan de manera coordinada y si uno de los extremos encuentra una secuencia homóloga, el otro extremo busca de manera más intensa en su vecindad. Sorprendentemente, algunas regiones del genoma se involucran más frecuentemente en la reparación de las roturas que otras, como el cromosoma III. Por lo tanto, el genoma no se explora de manera homogénea durante la búsqueda de homología, lo que sugiere la existencia de mecanismos de focalización que serán objeto de estudios posteriores.
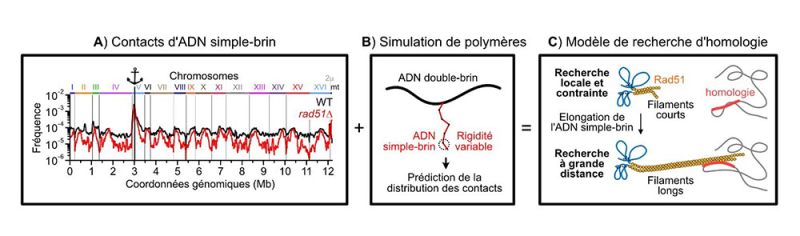
(A) La técnica ssHi-C permite determinar la frecuencia de contacto entre el ADN monocatenario generado a nivel de una rotura (representado por el ancla) y el resto del genoma en un contexto salvaje o mutante.
(B) Estos datos experimentales se comparan con las predicciones de simulaciones de polímeros.
(C) Lo que conduce a un modelo de expansión de la búsqueda de homología basado en la formación de largos filamentos rígidos formados por la proteína Rad51 a ambos lados de la rotura.
© Aurèle Piazza
Estos descubrimientos conducen a un modelo general de búsqueda de homología basado en la estructura rígida del filamento de Rad51 y sus socios. Abre nuevas direcciones de investigación, particularmente para entender los defectos asociados a mutaciones en genes supresores de tumores, como los parálogos de Rad51, en la regulación de la estructura del filamento de Rad51 y el alcance de la búsqueda de homología. Finalmente, la técnica ssHi-C, universalmente aplicable, debería permitir estudiar la localización espacial del ADN durante la replicación o su reparación en numerosos organismos y contextos celulares.
Referencias:
Mechanism of homology search expansion during recombinational DNA break repair in Saccharomyces cerevisiae.
A. Dumont, N. Mendiboure, J. Savocco, L. Anani, P. Moreau, A. Thierry, L. Modolo, D. Jost, A. Piazza.
Molecular Cell, 22 de agosto de 2024. DOI: 10.1016/j.molcel.2024.08.003