
Imagem de ilustração Pixabay
A estabilidade do material genético é essencial para o bom funcionamento de cada uma de nossas células. No entanto, acidentes podem ocorrer, por exemplo, sob o efeito de radiações ou erros durante a replicação do ADN. Para reparar essas quebras existem mecanismos extremamente conservados em todas as espécies que têm como função realizar uma reparação fiel.
A recombinação homóloga é uma via de reparação de alta fidelidade que utiliza uma molécula de ADN intacta de sequência idêntica como matriz. Essa sequência pode vir da cromátide irmã, do cromossomo homólogo mais distante, ou de outras sequências repetidas no genoma. Esta cópia homóloga intacta precisa ser identificada na vastidão do genoma e do espaço nuclear. Como essa busca pela agulha homóloga no palheiro do genoma é conduzida?
Para responder a essa questão, cientistas do CNRS desenvolveram uma técnica de genômica de alto rendimento chamada ssHi-C em levedura Saccharomyces cerevisiae, um modelo para o estudo dos mecanismos fundamentais de manutenção do ADN. Essa técnica permitiu mapear em escala genômica os contatos feitos pela quebra. Esses dados revelaram que a busca por homologia ocorre em duas fases principais:
- uma primeira fase na qual a estruturação do genoma pelas coesinas restringe a busca localmente, as coesinas sendo proteínas que garantem a coesão das cromátides irmãs, aprisionando o ADN em seu interior.
- uma segunda fase na qual a busca se liberta dessas restrições e investiga sítios genômicos distantes. Essa pesquisa de longo alcance é permitida pela produção de longos filamentos de ADN de fita simples ao redor da quebra e sua rigidez é aumentada pela formação de um filamento da recombinase Rad51. Essa proteína, que catalisa eventos de recombinação, atua nesse caso como uma vara de pescar, projetando sua linha feita de longos filamentos de ADN de fita simples longe no núcleo. As duas extremidades da quebra procuram de maneira coordenada, e se uma extremidade encontra uma sequência homóloga, a outra extremidade passa a procurar de forma mais intensa em sua vizinhança. De maneira surpreendente, algumas regiões do genoma engajam a quebra com mais frequência que outras, como o cromossomo III. O genoma, portanto, não é interpelado de maneira uniforme durante a busca por homologia, o que sugere a existência de mecanismos de direcionamento que serão objeto de estudos futuros.
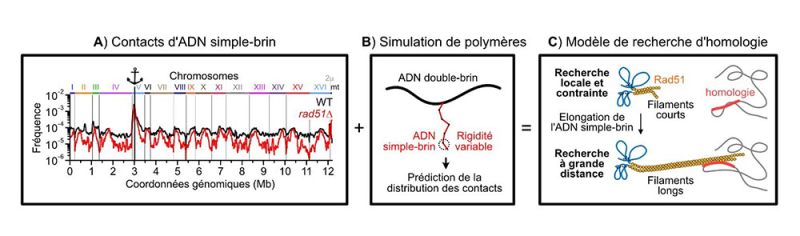
(A) A técnica ssHi-C permite determinar a frequência de contato entre o ADN de fita simples gerado em uma quebra (representado pela âncora) e o resto do genoma em um contexto selvagem ou mutante.
(B) Esses dados experimentais são comparados com previsões de simulações de polímeros.
(C) O que leva a um modelo de expansão da busca por homologia com base na formação de longos filamentos rígidos formados pela proteína Rad51 em ambos os lados da quebra.
© Aurèle Piazza
Essas descobertas conduzem a um modelo geral de busca por homologia baseado na estrutura rígida do filamento de Rad51 e seus parceiros. Elas abrem novas direções de pesquisa, notadamente para compreender as falhas associadas a mutações em genes supressores de tumor, como os paralogos de Rad51, na regulação da estrutura do filamento de Rad51 e no alcance da busca por homologia. Finalmente, a técnica ssHi-C, aplicável universalmente, deverá permitir o estudo da localização espacial do ADN durante a replicação ou sua reparação em diversos organismos e contextos celulares.
Referências:
Mechanism of homology search expansion during recombinational DNA break repair in Saccharomyces cerevisiae.
A. Dumont, N. Mendiboure, J. Savocco, L. Anani, P. Moreau, A. Thierry, L. Modolo, D. Jost, A. Piazza.
Molecular Cell, 22 de agosto de 2024. DOI: 10.1016/j.molcel.2024.08.003