Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
Des chercheurs du LCT (CNRS/Sorbonne Université), de l'IP2CT (CNRS/Sorbonne Université), de l'Université du Texas à Austin (États-Unis) et du GBMC (CNAM/HÉSAM Université) ont obtenu une simulation d'une protéine cruciale du virus responsable de la pandémie Covid-19 en deux semaines de calcul, alors que, à définition égale, les anciens procédés auraient pris une dizaine d'années. Ils ont pour cela adapté leurs codes à la simulation à champs de force polarisables, une méthode capable de mimer des phénomènes quantiques souvent négligés malgré leur importance dans la structure des protéines.
En parallèle de la publication de ces travaux dans la revue Chemical Science, l'ensemble des programmes informatiques impliqués ont été mis à disposition de la communauté scientifique afin de lutter contre l'épidémie.
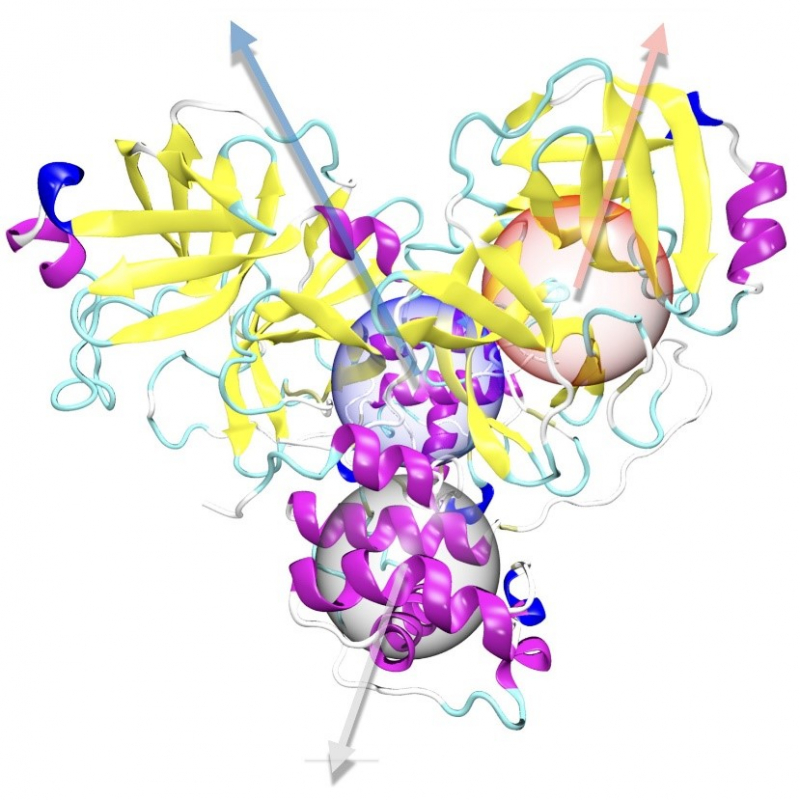
Modélisation de la protéase, les flèches indiquent trois sites d'intérêt étudiés lors de ces travaux. © Jaffrelot-Inizan et al. © Jean-Philip Piquemal
Afin de développer des traitements efficaces contre le Covid-19, des simulations ont été développées pour étudier la structure 3D de différentes protéines du virus SARS-CoV-2, ainsi que leur évolution sur de courtes échelles de temps. Un modèle réaliste de cette cible protéique permettrait de repérer des points faibles sur lesquels se focaliser, afin de trouver quelles molécules pharmaceutiques sont capables de les attaquer. Bien menée, cette approche est beaucoup plus rapide et efficace que s'il fallait passer au crible tous les composés disponibles: seules les molécules repérées comme prometteuses par la simulation sont alors testées.
La protéase principale du virus est une cible très importante car cette protéine, impliquée dans la réplication du virus, mute peu. Ainsi, tout médicament qui parviendrait à l'empêcher de fonctionner serait efficace contre l'ensemble des variants possibles du virus, comme c'est le cas de la trithérapie qui bloque la protéase du VIH. Mais pour simuler cette protéine, le modèle doit comprendre 100 000 atomes et gérer leurs interactions au sein de la molécule de la manière la plus réaliste possible pour être prédictif. Les simulations habituelles calculent ces phénomènes en ne prenant en compte que les comportements physiques et chimiques basés sur la physique classique. Les phénomènes quantiques ont pourtant un impact fort sur la structure des protéines, mais les introduire, même de façon approchée, complique tellement les calculs qu'il faudrait alors des dizaines d'années pour simuler la protéase principale de SARS-CoV-2 sur des supercalculateurs.
Des chercheurs du Laboratoire de chimie théorique (LCT, CNRS/Sorbonne Université), de l'Institut parisien de chimie physique et théorique (IP2CT, CNRS/Sorbonne Université), de l'Université du Texas à Austin (États-Unis) et du laboratoire Génomique, bioinformatique et chimie moléculaire (GBMC, CNAM/HÉSAM Université) sont cependant parvenus à obtenir une telle simulation en haute-définition en seulement deux semaines, sur le supercalculateur Jean Zay du CNRS.
Leur méthode, dite à champs de force polarisables, prend en compte les effets à n-corps, c'est-à-dire le phénomène quantique qui veut que le total des interactions entre plusieurs atomes soit différent de la somme de leurs interactions deux à deux. Ce phénomène est ici mimé en donnant à chaque atome la capacité de se déformer, en faisant en sorte qu'il ne soit plus considéré comme une simple sphère et reproduise ainsi mieux les fluctuations de ces interactions.
En plus de gagner en précision, l'approche théorique a été reformulée pour pouvoir utiliser efficacement le calcul haute performance (HPC). Cela a permis de modéliser le comportement de la protéase sur 40 microsecondes, soit 400 fois plus longtemps que les simulations généralement produites dans les études du domaine, qui nécessitent plusieurs mois de calcul. Ce saut technologique a été rendu possible en adaptant les codes de simulation à l'utilisation simultanée de centaines de processeurs graphiques (GPU), présents sur les supercalculateurs Jean Zay et Joliot Curie, du Très grand centre de calcul du CEA (TGCC).
Les données ainsi que le programme utilisé dans ces travaux, appelé Tinker-HP, ont été mis à la libre disposition de la communauté scientifique, afin de soutenir la recherche contre le Covid-19 (https://github.com/TinkerTools/tinker-hp). Un consortium public/privé s'est également monté avec les coauteurs et des laboratoires expérimentaux pour trouver, à partir de ces simulations, puis de synthétiser les meilleures molécules candidates capables de s'attaquer au virus.
Référence:
High-Resolution Mining of SARS-CoV-2 Main Protease Conformational Space: Supercomputer-Driven Unsupervised Adaptive Sampling
Theo Jaffrelot-Inizan, Frédéric Celerse, Olivier Adjoua, Dina El Ahdab, Luc-Henri Jolly, Chengwen Liu, Pengyu Ren, Matthieu Montes, Nathalie Lagarde, Louis Lagardère, Pierre Monmarché and Jean-Philip Piquemal Chem. Sci., 2021, Accepted Manuscript.
https://doi.org/10.1039/D1SC00145K
Contacts:
- Jean-Philip Piquemal - Chercheur, Laboratoire de chimie théorique (CNRS/Sorbonne Université) - jean-philip.piquemal at sorbonne-universite.fr
- Christophe Cartier dit Moulin - Chercheur à l'Institut parisien de chimie moléculaire & Chargé de mission pour la communication scientifique de l'INC - inc.communication at cnrs.fr
- Anne-Valérie Ruzette - Chargée scientifique pour la communication - Institut de chimie du CNRS - anne-valerie.ruzette at cnrs.fr
- Stéphanie Younès - Responsable Communication - Institut de chimie du CNRS - inc.communication at cnrs.fr