Prédire quelles protéines interagissent à partir de leurs séquences
Publié par Adrien,
Source: CNRS-INSBAutres langues:
Source: CNRS-INSBAutres langues:
1
Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
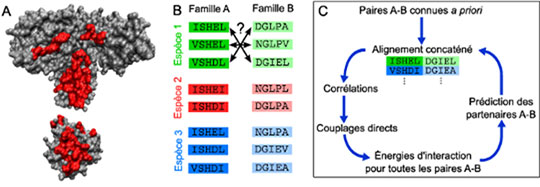
Figure: Détermination de partenaires d'interaction à partir des séquences. (A) Structure de deux protéines qui interagissent ; les zones d'interaction sont colorées en rouge. (B) On considère deux familles de protéines, constituées chacune de protéines homologues. Existe-t-il des interactions entre elles ? Si oui, quels membres de ces deux familles sont des partenaires d'interaction, au sein de chaque espèce ? (C) Grandes lignes de l'algorithme itératif proposé.
© Anne-Florence Bitbol
Les protéines assurent des fonctions cruciales dans chaque cellule vivante, comme la signalisation, le métabolisme ou la mobilité. Des interactions spécifiques entre différentes protéines régissent ces processus. Par exemple, dans les réseaux de signalisation, différentes protéines interagissent pour transmettre un signal et y répondre. Déterminer quelles protéines interagissent entre elles est donc crucial pour comprendre le fonctionnement cellulaire, et ouvre la porte au développement de nouveaux médicaments. Une résolution expérimentale complète de ce problème est difficile au vu du grand nombre de protéines différentes dans chaque cellule. Cependant, les séquences d'acides aminés constituant les protéines, c'est-à-dire leur composition chimique, sont de mieux en mieux connues. Peut-on identifier quelles protéines interagissent à partir uniquement de leurs séquences ?
Pour que deux protéines interagissent, elles doivent être complémentaires du point de vue physico-chimique. Deux familles de protéines qui interagissent entre elles ont donc des séquences corrélées. Les corrélations au sein des séquences sont une précieuse source d'information qui a déjà permis de déterminer la structure tri-dimensionnelle de certaines protéines à partir des séquences, résolvant ainsi le problème du repliement de ces protéines. Des chercheurs du laboratoire Jean Perrin, et des Universités de Princeton (États-Unis) et de Cambridge (Royaume-Uni) ont développé une approche qui exploite les corrélations entre séquences pour prédire quelles protéines interagissent entre elles parmi deux familles de protéines. Pour cela, une méthode d'inférence inspirée par la physique statistique et permettant de distinguer les corrélations indirectes et directes est utilisée de manière itérative. Un premier modèle est construit pour prédire les partenaires d'interaction, puis les meilleures de ces prédictions sont prises en compte afin de construire un nouveau modèle, améliorant les prédictions, et ainsi de suite. Cet algorithme itératif a été testé sur des protéines dont les partenaires d'interaction sont connus, et donne d'excellents résultats, même en ne supposant aucune connaissance a priori des interactions. Cette méthode rend possible la détermination d'interactions encore inconnues entre protéines à partir de leurs seules séquences.