Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
Les protéines ont la propriété d'être flexibles. La description précise de leur dynamique dans la cellule est très importante, notamment pour la compréhension de certaines de leurs fonctions clé, comme l'interaction avec d'autres molécules ou la catalyse de réactions chimiques. La spectroscopie par résonnance magnétique nucléaire (RMN) à l'état liquide est un outil bien établi pour examiner la structure et la dynamique des protéines en solution. Plus récemment, la RMN à l'état solide est apparue comme une nouvelle méthode, permettant cette fois-ci d'étudier des protéines à l'état solide, telles que les fibres amyloïdes ou les protéines membranaires. Elle apporte des informations inaccessibles à la diffraction aux rayons-X ou la RMN liquide.
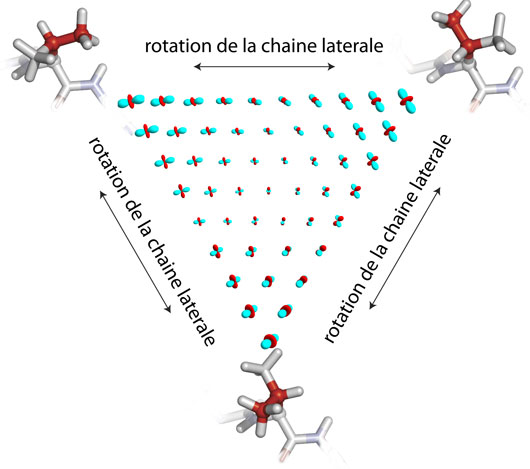
Figure: La chaîne latérale d'une protéine est dynamique et peut se retrouver sous trois états différents, représentés aux extrémités de la figure. Cette dynamique se traduit par une asymétrie du couplage dipolaire (symboles au centre) qui peut être mesurée par RMN du solide. Le mouvement de la protéine peut ainsi être identifié. © IBS, Paul Schanda
Toutes les techniques de RMN existant pour l'étude de la dynamique des protéines se fondent cependant sur un modèle assez simpliste, où l'amplitude de mouvement d'un site particulier de la protéine, comme une liaison chimique par exemple, est exprimée selon un paramètre unique, le "paramètre d'ordre". Elles ne rendent donc pas compte de l'asymétrie potentielle des mouvements moléculaires, qui sont particulièrement complexes au sein des protéines.
Les chercheurs de l'IBS et de l'ETHZ viennent de mettre au point une nouvelle expérience de RMN solide, qui permet de décortiquer plus précisément ces mouvements. Leur approche, basée sur un marquage isotopique (1) et une manipulation optimisée des spins (2), donne pour la première fois la possibilité de mesurer les couplages dipolaires (3) asymétriques dans les protéines. Ces couplages marquent la position des atomes les uns par rapport aux autres et reflètent directement le mouvement des liaisons au sein de la molécule.
Dans un premier temps, les scientifiques se sont intéressés à la dynamique des acides aminés portant un groupement méthyle. Cette dynamique est typiquement dominée par des transitions entre plusieurs états bien définis, appelés "rotamères" (voir figure). Grâce à la nouvelle approche développée, les chercheurs ont pu quantifier les populations relatives des différents rotamères dans une protéine modèle et discerner plus exactement l'asymétrie de ses mouvements. L'extension de cette méthode à des fibres amyloïdes ou des protéines membranaires permettra de mieux comprendre leur fonctionnement et leur stabilité, deux attributs quasiment insaisissables avec les autres méthodes qui sont utilisées en biologie structurale.
Notes:
(1) Le marquage isotopique consiste à remplacer tous les protons d'une molécule par du deutérium, sauf ceux qui se situent sur les chaines latérales des acides aminés que l'on veut étudier.
(2) Le spin est une propriété quantique qui caractérise chaque particule au même titre que sa masse et sa charge électrique. Pour observer la structure d'une molécule à un temps donné, la RMN utilise le phénomène de résonnance des spins nucléaires en présence d'un champ magnétique.
(3) Le couplage dipolaire fait référence à l'interaction, à travers l'espace, des spins nucléaires de deux atomes placés à proximité l'un de l'autre.
Référence:
Solid-state NMR measurements of asymmetric dipolar couplings provide insight into protein side-chain motion, Paul Schanda, Matthias Huber, Jerome Boisbouvier, Beat Meier Matthias Ernst, Angewandte chemie International edition 50(39):1-7, Published online before print September 14, 2011, doi:10.1002/anie.201103944.