Die Suche nach Arzneimittelkandidaten basiert hauptsächlich auf dem experimentellen oder numerischen Screening der größtmöglichen Anzahl von Molekülen, von denen angenommen wird, dass sie therapeutische Eigenschaften besitzen könnten. Bei einem experimentellen Screening bestimmt die Anzahl der für biologische Tests verfügbaren Moleküle den Bereich des Screenings, einen Bereich, der durch ein virtuelles Screening auf "theoretische" chemische Systeme erweitert werden kann.

Die Anzahl und die therapeutische Leistung der Moleküle, die biologische Tests erfolgreich bestehen können, hängen im Wesentlichen vom durchgescreenten "chemischen Raum" ab: Je größer die Molekülbank in Bezug auf Größe und chemische Vielfalt ist, desto effektiver ist das Screening.
Wissenschaftler des Labors für therapeutische Innovation (CNRS-Universität Straßburg) haben gerade besonders effektive Inhibitoren von therapeutisch relevanten Proteinen entwickelt, indem sie virtuell extrem breite theoretische chemische Räume (bis zu 5 Milliarden Moleküle) durchsuchten. Die von ihnen entwickelte Screening-Methode hat es ihnen ermöglicht, bisher fünfzehn Arzneimittelkandidaten zu identifizieren, deren Synthese und biologische Bewertung sie durchgeführt haben.
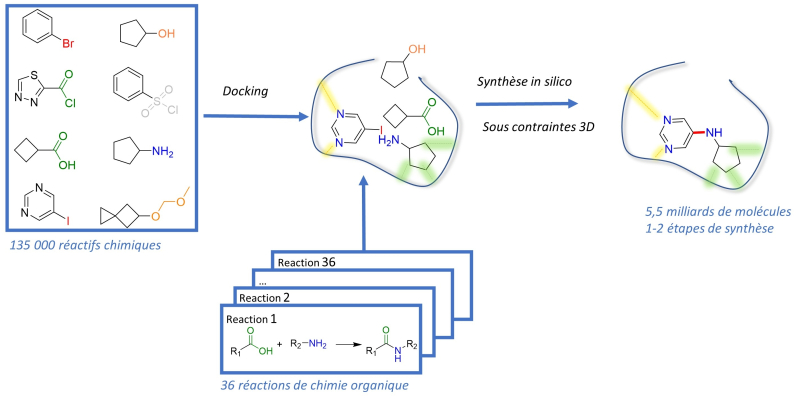
Diese innovative Herangehensweise, genannt "SpaceDock", beginnt mit der Identifizierung und Lokalisierung der aktiven Stellen des Proteins, mit denen die Inhibitoren interagieren könnten. Für den Dopamin D3-Rezeptor konnten so 135.000 kommerzielle chemische Reagenzien ausgewählt werden, die sich an dieser aktiven Stelle anlegen können.
Struktur eines Arzneimittelkandidaten gebunden an seinen Rezeptor, generiert durch künstliche Intelligenz
© Didier Rognan
Die Wissenschaftler berücksichtigten anschließend die topologischen Einschränkungen der dreidimensionalen Struktur des Proteins, um ihnen unter diesen Reagenzien jene zu zeigen, die nah genug kommen können, um zu interagieren und neue Moleküle zu formen... Hunderte Milliarden solcher Annäherungen sind möglich, deren Ergebnisse können durch die Anwendung von Verfahren für 40 „klassische“ Reaktionen der organischen Chemie vorhergesagt werden. Die größte jemals erstellte Screening-Bibliothek!
Diese Methode, die in der Zeitschrift ACS Cent. Sci., veröffentlicht wurde, ist ultraschnell, selektiv und zuverlässig und ermöglicht es, Moleküle mit therapeutischem Potential vorherzusagen, zu synthetisieren und dann zu testen, indem sie aus einer unvergleichlichen Palette chemischer Substanzen in Bezug auf Größe, Relevanz und Vielfalt schöpft.
Redakteur: CCdM
Referenz:
François Sindt, Anthony Seyller, Merveille Eguida & Didier Rognan.
Protein Structure-Based Organic Chemistry-Driven Ligand Design from Ultralarge Chemical Spaces
ACS Cent. Sci. 2024
https://pubs.acs.org/doi/10.1021/acscentsci.3c01521