Un mécanisme d'antibiorésistance inédit
Publié par Isabelle,
Source: INRAAutres langues:
Source: INRAAutres langues:
3
Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
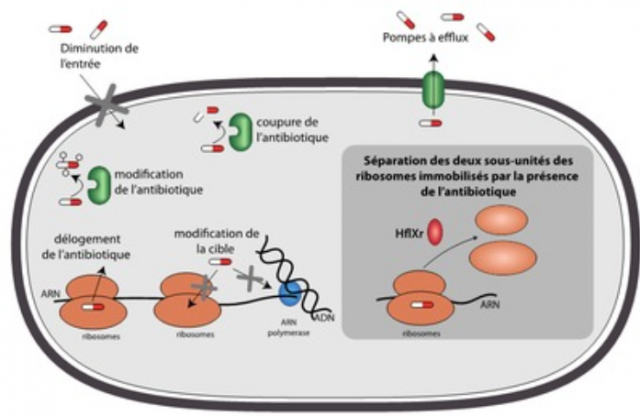
Les bactéries possèdent plusieurs mécanismes de résistance aux antibiotiques, tels que les pompes à efflux ou les enzymes de modification ou de coupure. HflXr, le gène découvert dans cette étude, permet le ré-amorçage de la synthèse protéique grâce à la dissociation des ribosomes bloqués.
© Institut Pasteur
Dans leur milieu naturel, les bactéries cohabitent avec une grande diversité de microorganismes. Pour survivre et accéder à leur nourriture, celles-ci n'ont d'autres choix que de repousser voire d'éliminer leurs concurrents en émettant des antibiotiques. Et il est donc aussi vital pour les bactéries de savoir s'en protéger. Ces dernières ont ainsi développé une palette très variée de mécanismes de défense contre les antibiotiques: certaines ont une membrane "imperméable" aux antibiotiques, d'autres pompent les antibiotiques vers l'extérieur, d'autres encore les modifient ou les coupent pour les rendre inactifs. "On a une vision assez claire des mécanismes de résistance mis en œuvre par les bactéries, mais, comme le montrent nos résultats, il y en a sans doute encore un certain nombre que l'on ne soupçonne pas", souligne Mélodie Duval, chercheur post-doctorante au sein de l'unité des Interactions bactéries-cellules de l'Institut Pasteur. "Il y a deux ans, en collaboration avec l'équipe israélienne de Rotem Sorek de l'Institut Weizmann des Sciences,nous avons identifié chez Listeria un premier gène de résistance induit par la présence d'antibiotiques(1), poursuit Pascale Cossart, directrice de l'unité et coordinatrice du Labex IBEID. La régulation de son expression était originale mais son mode d'action ne l'était pas. Cette fois-ci, le gène que nous venons de découvrir et caractériser induit un mécanisme de résistance totalement nouveau, qui me fascine", s'enthousiasme la chercheuse.
Tout débute, cette fois encore, avec la méthode d'analyse développée par l'équipe de Rotem Sorek(2). Baptisée "term-seq", cette technique permet de connaître, dans différentes conditions expérimentales, la longueur et l'abondance des ARN messagers (ARNm) dans un échantillon donné et donc le degré d'expression des gènes correspondants. "Nous avons testé l'effet de deux antibiotiques, la lincomycine et l'érythromycine, sur la bactérie Listeria monocytogenes", explique Mélodie Duval. Pour cela, les bactéries sont mises en culture, avec et sans antibiotique, puis leurs ARNm extraits. Résultat: certains ARNm, dont la transcription s'arrête prématurément en l'absence d'antibiotique, voient leur transcription se dérouler de manière complète en présence d'antibiotiques, dévoilant ainsi l'induction de certains gènes. "L'un d'entre eux a tout particulièrement attiré notre attention car il avait de fortes analogies avec un gène connu pour aider les bactéries E. coli à résister aux chocs thermiques, en séparant en deux parties leurs ribosomes bloqués, relate Mélodie Duval. Cela nous a semblé particulièrement intéressant puisque les antibiotiques utilisés dans notre étude, tout comme les chocs thermiques, sont connus pour bloquer les ribosomes des bactéries". Y avait-il une similarité de mode d'action ?
Effectivement, en étudiant ce gène de manière plus approfondie, les chercheurs s'aperçoivent que la protéine qu'il code agit au niveau des ribosomes – ces usines chargées de traduire l'ARNm en protéines – et le baptisent hflXr. Son expression, régulée par un mécanisme d'atténuation [3] et stimulée en présence d'antibiotique, permet de produire une protéine qui va littéralement séparer en deux les ribosomes. Loin de les détruire, cette action va permettre aux deux sous-unités ribosomiques d'être recyclées et de reprendre leur travail de synthèse protéique, un processus essentiel à la croissance bactérienne. "Cette séparation en deux des ribosomes pour résister à un antibiotique est un mécanisme totalement nouveau", affirme Pascale Cossart. Un mécanisme de résistance qui ne semblerait d'ailleurs pas être l'exclusivité de Listeria puisque le gèneh flXr, et potentiellement le mécanisme associé, est présent chez un grand nombre de bactéries, tout particulièrement chez les firmicutes. "Parallèlement à nos travaux, des scientifiques croates et canadiens qui recherchaient des gènes de résistance aux antibiotiques dans des échantillons de sol, prélevés à proximité d'une usine de fabrication d'antibiotiques et d'une ferme d'élevage, ont mis en évidence la présence du gène hflXr(3,4). C'est gratifiant de savoir que des chercheurs ont détecté, dans l'environnement, ce que nous avons découvert et disséqué chez Listeria en laboratoire. Cela renforce de façon élégante les résultats de notre travail fondamental", conclut Mélodie Duval.
Ce travail a reçu des financements de l'institut Pasteur, l'Inserm, l'Inra, le CNRS, l'ERC (ADV Grant BacCellEpi), l'Institut Weizmann Pasteur, la Fondation Le Roch Les mousquetaires ainsi que le Labex IBEID.
Notes:
(1) Dar D, Shamir M, Mellin JR, et al (2016) Term-seq reveals abundant ribo-regulation of antibiotics resistance in bacteria. Science 352:1–12 . doi: 10.1126/science.aad9822
(2) Duval M, Dar D, Carvalho F, Rocha E.P.C., Sorek R and Cossart P, HflXr, an homolog of a ribosome-splitting factor, mediates antibiotic resistance.Proc. Natl. Acad. Sci USA 2018, in press
(3) mécanisme de régulation de l'expression des gènes présent chez les bactéries, où la transcription se termine de façon prématurée, en amont du gène de structure, et où il n'y a donc pas d'expression.
(4) González-Plaza JJ, Šimatovic A, Milakovic M, et al (2018) Functional Repertoire of Antibiotic Resistance Genes in Antibiotic Manufacturing Effluents and Receiving Freshwater Sediments. Front Microbiol 8:1–13 . doi: 10.3389/fmicb.2017.02675
(5) Lau CH-F, Van Engelen K, Gordon S, et al (2017) Novel antibiotic resistant gene from agricultural soil exposed to antibiotics widely used in human medicine and animal farming. Appl Environ Microbiol 83:1–18 . doi: 10.1128/AEM.00989-17
Références pbblication:
Mélodie Duval, Daniel Dar, Filipe Carvalho, Eduardo P. C. Rocha, Rotem Sorek, and Pascale Cossart HflXr, a homolog of a ribosome-splitting factor, mediates antibiotic resistance, PNAS, 13 decembre 2018