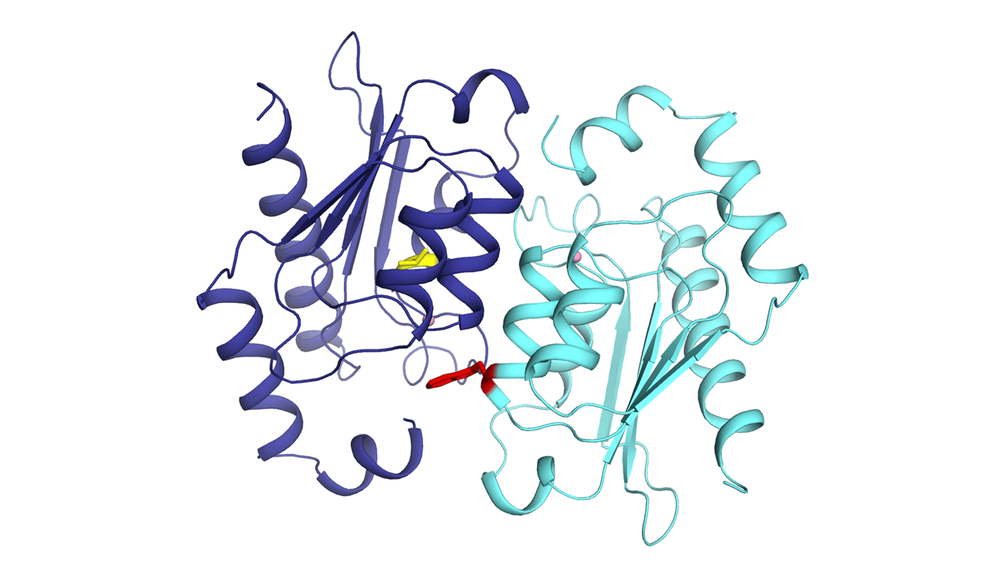
Zwei Kopien eines Bäckerhefe-Proteins in einer Abhängigkeitsbeziehung. Funktionsverlust-Mutationen (in Gelb und Rot) machen sie ohne Zusammenarbeit funktionsunfähig.
Philippe Després
Für eine identische zelluläre Funktion enthält die Proteinmaschine des Menschen oft mehr Komponenten als die einer Bakterie. „Man würde annehmen, dass diese Komponenten durch die natürliche Selektion hinzugefügt wurden, dass die Komplexität einen Nutzen bringt oder die Leistung beim Menschen verbessert. Aber das ist nicht unbedingt der Fall. Die Maschine der Bakterie ist oft effizienter, auch mit weniger Bauteilen“, betont Philippe Després.
Obwohl die Komplexität keinen unmittelbaren negativen Effekt auf die Effizienz hat, müssen die hinzugefügten Komponenten dennoch produziert werden, was mehr Energie erfordert. Dadurch wird die Maschine schwieriger zu erhalten, erklärt der junge Forscher, der seine Doktorarbeit unter der Leitung von Professor Christian Landry verfasst hat.
Eine „toxische“ Beziehung
Wenn es keinen Mehrwert gibt, welche Mechanismen erklären dann die Komplexität der Proteinmaschinen beim Menschen? Ein vor etwa zwanzig Jahren entwickeltes theoretisches Modell legt nahe, dass zufällige Mutationen die Ursache sein könnten.
„Irgendwann in der Evolution wurde das Gen, das eines der Proteine der Maschine kodiert, dupliziert, und es durchlief anschließend Funktionsverlust-Mutationen. Die neuen, von diesen mutierten Genen kodierten Proteine entwickelten eine Abhängigkeitsbeziehung, eine toxische Beziehung. Anders ausgedrückt: Die beiden Kopien der Proteine müssen sich zur Erfüllung derselben Funktion wie die ursprüngliche einzelne Kopie zusammenschließen“, erklärt Philippe Després.
Dem jungen Forscher zufolge könnte diese Theorie erklären, warum die Protein-Komplexe des Menschen mit der Zeit tendenziell immer größer werden. „Dieser zufällige Mechanismus wirkt gewissermaßen gegen die natürliche Selektion“, fügt er hinzu.
Ein Machbarkeitsbeweis
Dieses theoretische Modell der Komplexitätsbildung wurde bisher nicht im Labor nachgewiesen. Um dies zu erreichen, verwendete das Team ein Protein der Bäckerhefe als Modell. Die Forscher setzten es Mutationen aus und stellten fest, dass mehrere Kombinationen zur Abhängigkeit und Komplexitätssteigerung führten.
Weniger als 1 % der Kombinationen entwickelten eine Abhängigkeitsbeziehung, um funktionieren zu können, ein höherer Prozentsatz, als das Team erwartet hatte. „Das klingt nach wenig“, räumt der Forscher ein. „Aber auf evolutionärer Zeitskala ergeben sich viele Gelegenheiten für Komplexitätsereignisse. Der Prozess könnte weit verbreiteter sein, als wir annehmen.“
Die Idee der Abhängigkeit in Proteinmaschinen könnte wichtige Implikationen für die Krebsforschung haben. „In Krebszellen können die beiden Kopien unabhängig voneinander mutieren. Früher, wenn beide Kopien Funktionsverluste erlitten, ging man davon aus, dass das Gen nicht mehr funktionsfähig war. Jetzt müssen wir prüfen, ob diese Kopien sich immer noch zusammenschließen und eine funktionale Maschine bilden können“, erklärt Philippe Després, der nun als Postdoktorand am Broad Institute in Boston und an der Universität Guelph in Ontario tätig ist.
Das „Highlight“, so der Forscher, bestand darin, die molekulare Struktur der Proteine bis auf das Atom genau zu analysieren. „Das ist eine beispiellose Auflösung, um zu untersuchen, wie sich die beiden Proteine trotz der Funktionsverlust-Mutationen zusammenschließen, um zu funktionieren.“ Er betont die Zusammenarbeit mit dem Team von Professor Rong Shi vom Fachbereich für Biochemie, Mikrobiologie und Bioinformatik der Universität Laval.
Zu den weiteren Mitautoren der in der Zeitschrift Science veröffentlichten Studie gehören Alexandre K. Dubé, Marie-Ève Picard, Jordan Grenier, Rong Shi und Christian Landry.