Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
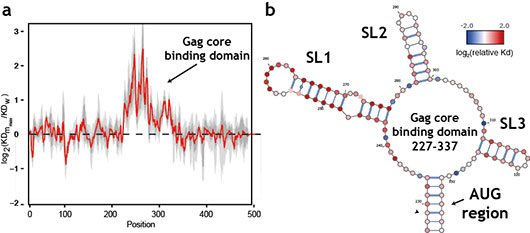
Figure: Analyse de la fixation du précurseur des protéines structurales Pr55Gag à l'ARN du VIH-1 par MIME.
(a) Effet des mutations sur la constante d'affinité des complexes.
(b) Les données sont reportées sur la structure du site de fixation.
© Roland Marquet. Redmond P. Smyth
Les multiples fonctions de l'ARN reposent sur sa capacité à former des structures tridimensionnelles complexes qui interagissent de manière spécifique avec des protéines, des acides nucléiques ou de petits ligands. Traditionnellement, l'étude des relations structure/fonction des ARN repose sur une approche pas à pas requérant l'identification d'un domaine fonctionnel, sa caractérisation structurale, et l'identification des résidus cruciaux par l'analyse séquentielle de multiples variants obtenus par mutagenèse dirigée.
La méthodologie développée par les chercheurs strasbourgeois et leurs collaborateurs consiste à introduire des mutations ponctuelles aléatoires dans l'ARN, à séparer les molécules d'ARN actives pour la fonction étudiée (ici la fixation à une protéine) des molécules inactives et à séquencer les mutations présentes dans chacune des populations par séquençage à haute débit. Cette approche, appelée MIME (Mutational interference mapping experiment) permet d'étudier l'effet de plusieurs milliers de mutations simples ou centaines de milliers de mutations doubles dans une seule expérience. Son originalité repose sur l'introduction d'une étape de fragmentation avant l'étape de séquençage pour s'affranchir de la longueur limitée des séquences obtenues à haut débit, et sur le développement d'outils mathématiques permettant d'extraire des paramètres quantitatifs et une analyse statistique robuste directement à partir des données de séquençage. La méthode MIME permet non seulement d'identifier les nucléotides importants pour la fonction, mais aussi de mettre en évidence des motifs structuraux fonctionnels de l'ARN grâce à l'analyse des effets des différentes substitutions à chaque position et à l'étude de l'effet de paires de substitutions.
Dans leur étude princeps, les chercheurs ont validé la méthode MIME en montrant qu'elle permet d'identifier le site, bien connu, de liaison de la protéine de capside du phage MS2 à l'ARN, puis ils l'ont utilisée pour étudier la fixation du précurseur Pr55Gag à l'ARN génomique du VIH-1. Cette interaction permet le recrutement de l'ARN au sein des nouvelles particules virales produites par les cellules infectées. La région de l'ARN génomique requise pour la fixation de Pr55Gag s'étend sur plus de 100 nucléotides et les données de MIME ont permis de trancher entre plusieurs modèles structuraux alternatifs de cette région proposés dans la littérature.
La méthode MIME n'est pas restreinte à l'étude des interactions ARN/protéine car elle peut être appliquée à des processus très variés, aussi bien in vitro qu'en culture cellulaire, la seule limitation étant que les ARN fonctionnels et non fonctionnels puissent être séparés physiquement.