ProteoRE, une application web pour la découverte de biomarqueurs d'intérêt diagnostique
Publié par Redbran,
Source: Institut de recherche interdisciplinaire de Grenoble (lRlG)Autres langues:
Source: Institut de recherche interdisciplinaire de Grenoble (lRlG)Autres langues:
Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)

Un biomarqueur est une caractéristique mesurable avec précision, utilisée comme indicateur d'une fonction du corps, d'une maladie ou de l'action d'un médicament. À titre d'exemple, la glycémie est un biomarqueur reconnu tant pour caractériser le diabète que pour évaluer l'efficacité des molécules antidiabétiques. Dans le domaine médical, un biomarqueur peut être utilisé pour le dépistage, le diagnostic, la réponse à un traitement, la rechute après un traitement, etc. Un biomarqueur peut être une molécule (métabolites, ADN circulants, protéines, etc.) facilement mesurable dans le sang ou dans l'urine et compatible avec une analyse en routine. Le développement d'un biomarqueur, de sa découverte à sa validation, est un processus long et coûteux: comment accélérer le processus de découverte de nouveaux candidats biomarqueurs à des fins d'évaluation en recherche clinique ?
Bien que les biomarqueurs soient largement utilisés, des limites importantes ont été identifiées en raison de leur manque de spécificité ; par exemple en cancérologie, l'absence de marqueur unique hautement spécifique et sensible a conduit à un consensus croissant sur la nécessité de combinaisons de biomarqueurs multiparamétriques. Une approche fondée sur une connaissance a priori (i.e. reposant sur des critères mécanistiques propres à la genèse d'une pathologie) tirant partie des bases de données "omiques" sans cesse grandissantes, pourrait constituer un moyen puissant d'identifier des biomarqueurs, en proposant aux chercheurs biologistes – pas forcément experts en programmation ou en bio-informatique – des moyens informatiques puissants pour recueillir les informations pertinentes dans la masse croissante de données disponibles.
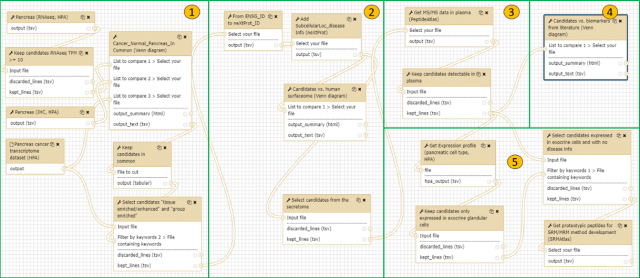
Sur la base de ce constat, des chercheurs de notre institut ont conçu ProteoRE, une application Web proposant un ensemble d'outils accessibles aux biologistes afin de définir leur propre stratégie de sélection de candidats biomarqueurs. Ces outils reposent sur l'extraction de données expérimentales à partir de bases de données (protéomique et transcriptomique) publiques humaines et l'application de filtres successifs basés sur des critères physiologiques et biochimiques (e.g. spécificité tissulaire, localisation subcellulaire, détectabilité, etc.) en relation avec la pathologie. Un protocole étape par étape, qu'on appelle "workflow", permet de relier ces outils de façon transparente et reproductible (Figure). L'exécution de ce type de workflow via ProteoRE a ainsi permis a ces chercheurs de sélectionner des candidats spécifiques du muscle cardiaque, localisés dans le cytoplasme des cellules, détectables et marqueurs potentiels de lésions du tissu cardiaque [1]. Dans une autre étude visant à élargir la liste de biomarqueurs candidats au diagnostic précoce du cancer du pancréas, cette stratégie a permis d'établir une liste de 14 biomarqueurs candidats [2] dont l'efficacité de certains a été corroborée par des études réalisés sur des patients diagnostiqués à un stade précoce - pour évaluation ultérieure par protéomique ciblée.
Les outils bio-informatiques, les workflows, résultats et tutoriel issus de ces stratégies sont mis à la disposition des chercheurs pour qu'ils puissent les réutiliser dans leur quête de découverte de biomarqueurs.
Projet ProteoRE
Le projet ProteoRE (Proteomics Research Environment) est un projet national financé par l'Institut français de bio-informatique (IFB – ANR-11-INBS-0013), en collaboration avec l'INRA (plate-forme MIGALE) et réalisé en coopération avec l'infrastructure protéomique (ProFI). ProteoRE est une application Web basée sur Galaxy ouvert à la communauté et dédiée à l'analyse fonctionnelle et l'exploration des données protéomiques et transcriptomiques dans la recherche biomédicale. Cet environnement permet aux chercheurs non-bioinformaticiens d'appliquer une large gamme d'outils ainsi que des workflows d'analyse sur leurs données, de partager leurs résultats avec leurs collaborateurs, et de reproduire la même analyse en gardant trace du processus global. Actuellement, ProteoRE comprend 20 outils permettant la manipulation des données, l'annotation, la classification, les analyses d'enrichissement et construction de réseaux biologiques, avec les visualisations associées.
Approches "omiques"
Les approches "omiques" dont caractérisées comme étant des techniques à haut débit permettant une analyse simultanée d'un grand nombre de variables, elles comprennent principalement: la génomique, la transcriptomique (expression des gènes et leur régulation), la protéomique (analyse des protéines), la métabolomique (étude des métabolites produits). Ces approches permettent d'obtenir de très nombreuses informations sur la réponse cellulaire et/ou tissulaire à une exposition in vitro ou in vivo.
Voir aussi le site du projet ProteoRE: http://www.proteore.org/
Notes:
[1] Nguyen L, Brun V, Combes F, Loux V and Vandenbrouck Y. Designing an in silico strategy to select tissue-leakage biomarkers using the galaxy framework. Methods in Molecular Biology, 2019
[2] Vandenbrouck Y, Christiany D, Combes F, Loux V and Brun V. Bioinformatics tools and workflow to select blood biomarkers for early cancer diagnosis: An application to pancreatic cancer. Proteomics, 2019