Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
Cette percée scientifique majeure, fruit d'une collaboration internationale au sein du 4D Nucleome Project, dévoile l'organisation tridimensionnelle et dynamique du génome humain. En étudiant des cellules souches embryonnaires et des fibroblastes, les chercheurs ont capturé les principes qui régissent la manière dont les chromosomes se plient et interagissent dans l'espace confiné du noyau, influençant directement l'activité des gènes.

L'architecture intime du noyau
Contrairement à l'image linéaire héritée du séquençage, l'ADN adopte une conformation spatiale extrêmement organisée. Il forme des boucles, des domaines et des compartiments dont la géométrie conditionne l'accès aux machines moléculaires chargées de lire l'information génétique. Cette structure en 3D n'est pas aléatoire ; elle détermine quels gènes sont activés ou réprimés, sculptant ainsi l'identité et la fonction de chaque cellule.
Pour cartographier ce paysage, les scientifiques ont fusionné les données de plusieurs technologies génomiques de pointe. Cette approche intégrative a permis de dépasser les limites de chaque méthode isolée. Le résultat est un ensemble de modèles à haute résolution qui capturent la variabilité architecturale d'une cellule à l'autre, offrant une vision plus nuancée et réaliste de la vie intranucléaire.
Le travail, publié dans Nature, a identifié plus de 140 000 interactions sous forme de boucles chromatiniennes par type cellulaire. Ces boucles rapprochent physiquement des séquences d'ADN éloignées, comme des interrupteurs génétiques avec les gènes qu'ils contrôlent. La carte précise de ces points d'ancrage moléculaires est une clé pour comprendre les mécanismes fondamentaux de la régulation génique.
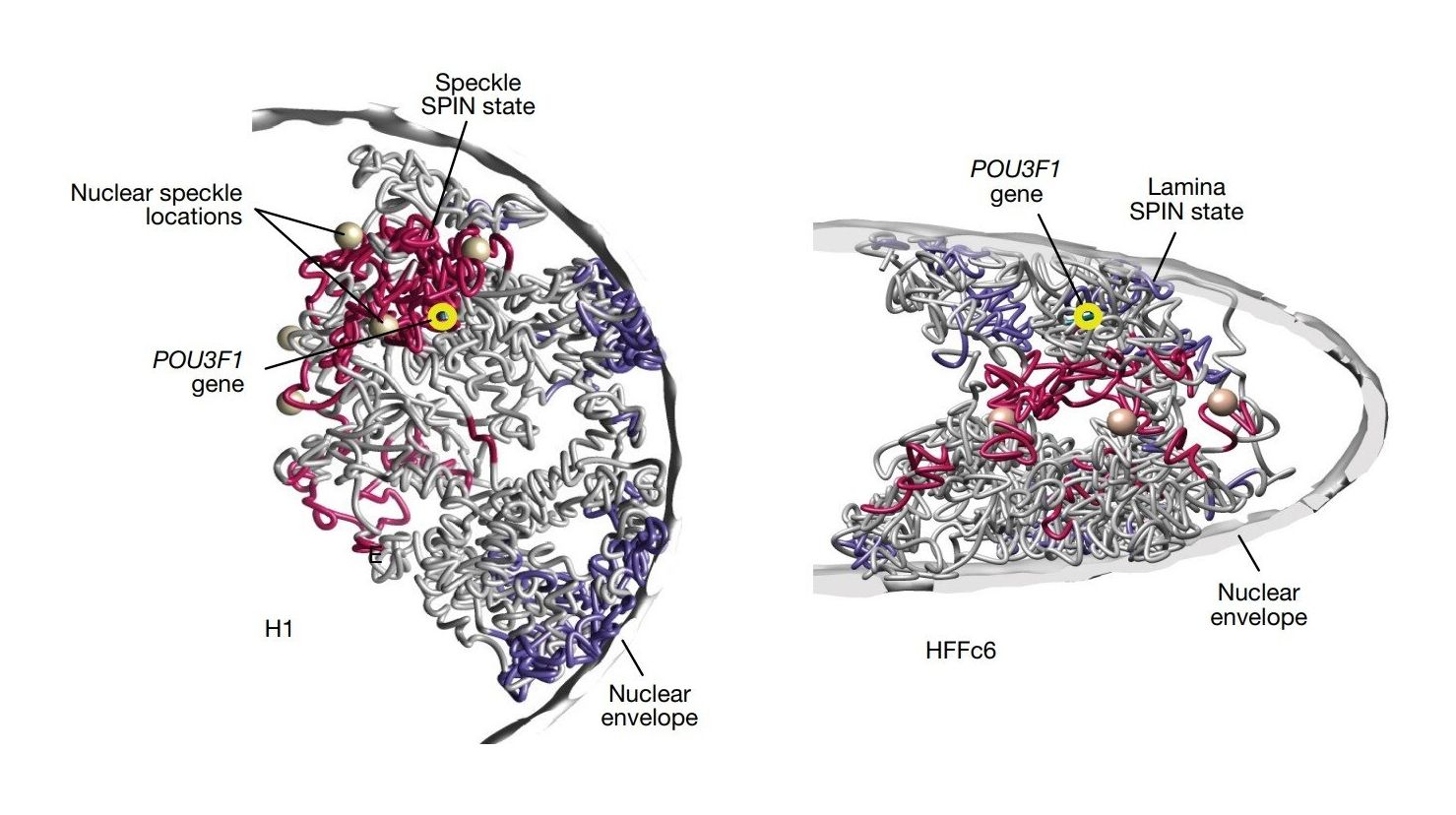
Structures 3D unicellulaires représentatives du chromosome 1 dans les cellules H1 (à gauche) et HFFc6 (à droite).
Crédit: Nature (2025)
Prédire les effets des mutations par la forme
L'un des apports les plus significatifs de cette étude réside dans le développement d'outils computationnels capables de prédire la façon dont une séquence d'ADN va se replier. Cette avancée ouvre la perspective de prévoir l'impact de variations génétiques sur la structure du génome sans recourir à des expériences en laboratoire. Elle est essentielle pour interpréter les mutations trouvées dans les régions non codantes, souvent impliquées dans les maladies.
En effet, de nombreuses variations associées à des pathologies comme le cancer ou des troubles du développement ne modifient pas directement un gène, mais perturbent probablement son environnement structural en 3D. La nouvelle cartographie fournit un cadre pour identifier quels gènes pourraient être affectés par ces altérations à distance, reliant ainsi des mutations cryptiques à leurs conséquences biologiques.
Les chercheurs ont également établi un guide méthodologique rigoureux en comparant les performances des différentes techniques de cartographie génomique. Ce référentiel permet à la communauté scientifique de choisir les outils les mieux adaptés pour étudier un aspect spécifique de l'organisation nucléaire, qu'il s'agisse des boucles, des domaines chromosomiques ou du positionnement dans le noyau.
La finalité de ces travaux est translationnelle. En ayant observé des anomalies structurales du génome dans des leucémies ou des tumeurs cérébrales, l'équipe vise maintenant à explorer comment cibler pharmacologiquement cette architecture, par exemple avec des inhibiteurs épigénétiques. Comprendre la forme du génome pourrait ainsi déboucher sur de nouvelles stratégies diagnostiques et thérapeutiques.