Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
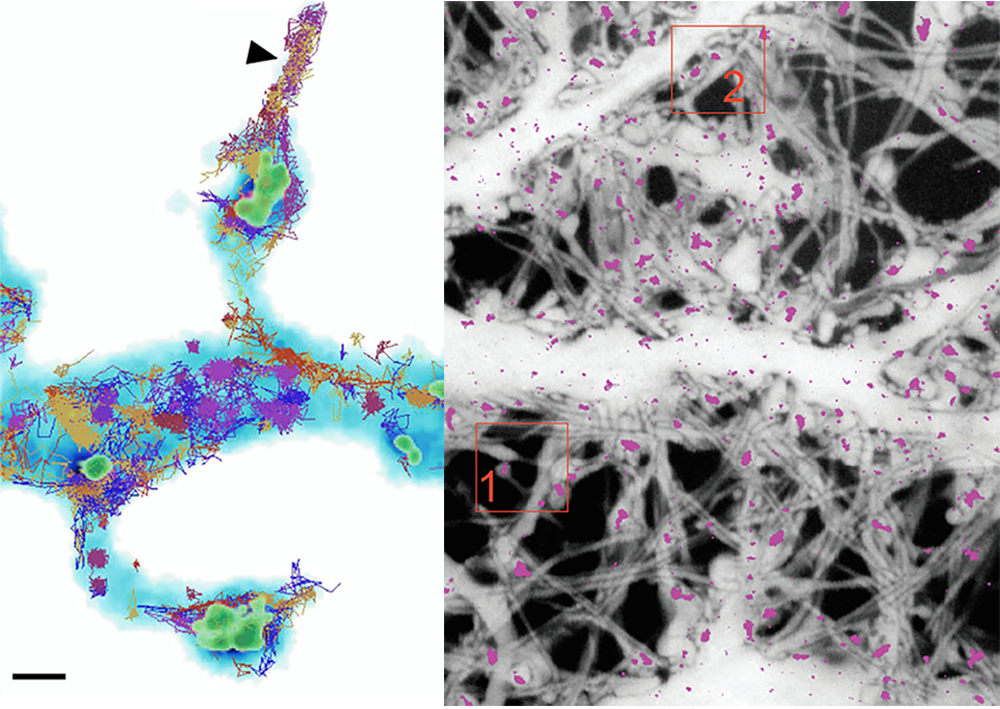
Figure: Super-résolution au carré: L'image de gauche montre les positions et les mouvements de protéines d'échafaudages et protéines de signalisation (taches vertes et traces ondoyantes) à l'intérieur et autour des épines dendritiques (bleu clair), qui sont le site des synapses excitatrices des neurones. Sur l'image de droite, le marquage ponctuel montre la localisation nanoscopique des synapses au sein de la morphologie complexe et dense des neurones.
© Valentin Nägerl & Jean-Baptiste Sibarita
La microscopie de super-résolution, ou nanoscopie, est devenue un outil de recherche important pour la biologie cellulaire et les neurosciences. Elle permet l'accès optique aux compartiments nanométriques (1 nanomètre = 1 milliardième de mètre) et à la dynamique de signalisation à l'intérieur des cellules vivantes. Au cours des dernières années, nous avons assisté à une diversification et à une cross-fertilisation importantes de ces techniques, permettant l'observation et l'analyse de plus en plus précise d'échantillons biologiques complexes. Toutefois, les deux principales techniques de super-résolution, fondées sur l'induction de fluorescence stochastique (par la localisation de molécules individuelles) ou déterministe (par déplétion), ont évoluées séparément en tant que technologies concurrentes, laissant leur synergie potentielle inexploitée. Compte tenu de leur complémentarité, leur combinaison au sein d'un même microscope pourrait offrir une solution "best of two-worlds", comblant ainsi le fossé des connaissances entre l'organisation moléculaire et morphologique des cellules.
Les chercheurs ont développé un tel microscope, permettant d'observer à l'échelle nanométrique les positions et mouvements des protéines synaptiques dans le contexte morphologique de la synapse. Ils démontrent ainsi la performance supérieure du nouveau système par rapport aux approches existantes, en offrant l'accès à la connaissance synchrone de ces deux informations cruciales pour déchiffrer la nanobiologie cellulaire.
Ce nouvel instrument a été conçu de manière modulaire et polyvalente, permettant aisément d'intégrer d'autres techniques de nanoscopie de pointe, telles que le uPAINT (Universal Point Accumulation for Imaging Nanoscale Topography) et le SUSHI (SUper-resolution Shadow Imaging), mises au point ces dernières années.
De nouvelles fonctionnalités qui pousseront les performances du système à un niveau encore plus élevé, tout en le rendant plus facile d'utilisation, sont déjà en cours de développement. Cela devrait ouvrir la voie à de nouvelles découvertes en biologie cellulaire et neurosciences.
Pour en savoir plus
A super-resolution platform for correlative live single-molecule imaging and STED microscopy.
Inavalli VVGK, Lenz MO, Butler C, Angibaud J, Compans B, Levet F, Tønnesen J, Rossier O, Giannone G, Thoumine O, Hosy E, Choquet D, Sibarita JB, Nägerl UV.
Nat Methods. 2019 Oct 21. doi: 10.1038/s41592-019-0611-8. [Epub ahead of print]