Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)

© Human Proteome Organization
En 2010, à l'occasion de son congrès annuel qui s'était tenu à Sidney (Australie), la Human Proteome Organization (HUPO ; www.hupo.org), a donné le top départ du Human Proteome Project (HPP). Il s'agit d'un projet mondial qui vise à caractériser l'ensemble des protéines codées par le génome humain. Dans ce contexte, chaque pays a adopté un chromosome (le chromosome 14 pour la France). Cet effort international de caractérisation précise et à grande échelle des protéines dans divers organes, cellules et fluides biologiques humains n'aurait pu atteindre ses objectifs sans un partage de données entre les "Chromosome teams", la définition et l'adoption de Guidelines très strictes en spectrométrie de masse et un environnement Assurance Qualité.
Au cours des dix dernières années, les équipes de recherche impliquées dans ce projet ont pu s'appuyer sur des ressources clés du HPP que sont les bases de connaissance Human Protein Atlas (Suède), PeptideAtlas et MassIVE (USA) et neXtProt (Suisse). Une analyse puis ré-analyse très rigoureuse des données produites par la communauté internationale en protéomique a permis de caractériser à plus de 90% le protéome humain. Les gènes sont un support d'information et n'ont à ce titre aucune fonction biologique. En revanche, ils codent pour des protéines qui elles portent des fonctions et jouent un rôle essentiel dans la physiologie. Les connaissances acquises dans le cadre du HPP sont essentielles pour discerner le rôle du protéome dans la santé et les pathologies.
La contribution française (Chromosome team 14) a été significative avec en particulier la caractérisation de près de 300 Missing proteins à partir d'échantillons de spermatozoïdes humains. Elle implique les ressources et savoir-faire de l'infrastructure nationale de protéomique ProFi (Grenoble, Strasbourg, Toulouse) et de la plate-forme Protim (Rennes).
La mission du Human Proteome Project est de ré-analyser et d'intégrer les données protéomiques produites par la communauté spécialiste du domaine selon des processus particulièrement rigoureux, apportant ainsi une meilleure compréhension moléculaire de la nature dynamique du protéome, incluant toutes ses modifications physiologiques et ses implications dans les processus pathologiques. Cette mission s'aligne avec la devise de HUPO "traduire le code de la vie", en fournissant des informations cruciales que la génomique seule ne peut pas fournir. L'achèvement du Human Proteome Project améliorera notre compréhension de la biologie moléculaire et cellulaire humaine au service de la médecine du futur et des patients. Ces travaux internationaux servent de base à des applications diagnostiques, pronostiques, thérapeutiques et sont le préambule indispensable au développement de la médecine de précision.
En parallèle de cet événement et pour illustrer les innovations et progrès majeurs accomplis par la communauté scientifique, HUPO a créé une vidéo retraçant de façon chronologique les évènements marquants dans le domaine de la protéomique. Elle est accessible aux chercheurs et au grand public (www.hupo.org/Proteomics-Timeline).
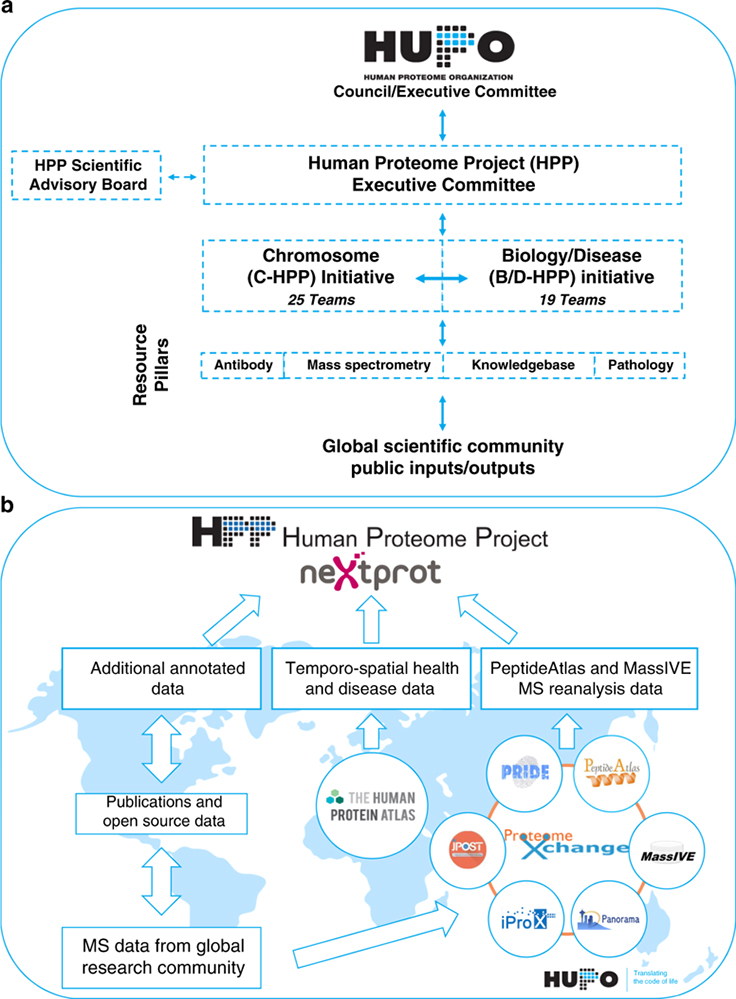
Figure : Architecture du Human Proteome Project.
a. Deux initiatives majeures complémentaires (C-HPP et B / D-HPP) impliquant de nombreuses équipes internationales qui s'appuient sur 4 piliers de ressources partagées (Anticorps, Spectrométrie de masse, Bases de données et Pathologie).
b. Les ressources partagées dans le pipeline HPP permettent l'intégration des données biologiques collectées qui sont alors traitées, ré-analysées et présentées chaque année.
Ces données sont accessibles à l'ensemble de la communauté scientifique selon le principe du FAIR data. Les jeux de données de spectrométrie de masse sont déposés, étiquetés avec un identifiant PXD et stockés selon différents référentiels PX (PRIDE, PeptideAtlas, MassIVE, Panorama, iProX, JPOST). La sélection, l'extraction et la ré-analyse des données par PeptideAtlas et MassIVE aboutissent à des informations de haute valeur ajoutée qui sont transmises à neXtProt. Par la suite, neXtProt agrège et intègre d'autres données biologiques (séquençage de Sanger, interaction protéine/protéine, données structurelles/cristallographiques) et l'ensemble est diffusé à la communauté scientifique de façon régulière via une version annuelle de référence (exemple: neXtProt release 17-01-2020).
© Adhikari et al.
Pour en savoir plus:
A high-stringency blueprint of the human proteome
Adhikari et al.,.
Nature Commmunications 16 Oct 2020. doi.org/10.1038/s41467-020-19045-9.
Laboratoire:
Biologie Santé et Innovation Technologique (BIOSIT)- (CNRS/ Inserm/ Université Rennes 1)
2, Avenue du Pr Léon Bernard, Campus Santé Villejean, 35043 Rennes cedex.
Contact:
- Charles Pineau - Directeur de recherche INSERM au Biologie santé et innovation technologique - [email protected]