Réplication de l'ADN - Définition
La liste des auteurs de cet article est disponible ici.
La fourche de réplication
La fourche de réplication est la structure formée lorsque l’ADN se réplique, et sur lesquelles l'ADN polymérase vient se fixer. L'ADN polymérase est une enzyme catalysant la formation des liaisons nucléotidiques. Le complexe enzymatique intervenant dans la réplication est appelé réplicase.
La réplication peut être divisée en trois étapes principales : l’initiation, l’élongation et la terminaison.
L’initiation de la réplication
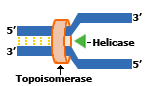
L’initiation de la réplication a lieu à l’origine de réplication. Il n’y a qu’une seule origine de réplication dans les chromosomes bactériens alors qu’il en existe plusieurs chez les eucaryotes. (Chez les Archaea, le nombre d’origine varie entre une et trois selon les espèces.) Il existe des protéines capables de reconnaître ces origines et initier la réplication. Ces protéines sont capables d’ouvrir progressivement et dérouler l’ADN. La fourche de réplication est créée par l’action des hélicases qui brisent les liaisons hydrogènes entre les deux brins de la double hélice d’ADN ce qui permet leur séparation. D’autres protéines peuvent se lier à l’ADN simple brin (ou ADN monocaténaire) ainsi formé et éviter la reformation de la double hélice. Ce sont les protéines SSB (single strand binding) des bactéries et les protéines RPA des eucaryotes et des archées.
L’élongation ou la synthèse d’ADN
C’est au cours de cette phase qu’il y a formation du réplisome et synthèse d’ADN. L’élongation de l’ADN progresse toujours dans le sens 5' vers 3' pour le brin en création. C’est l'ADN polymérase, qui ajoute à l'extrémité 3' de la molécule en formation, des désoxyribonucléotides. Cependant, les deux brins de la double hélice d’ADN sont enroulés dans des sens opposés : ils sont antiparallèles. Il existe de ce fait des mécanismes différents selon le brin d’ADN répliqué.
Il existe ainsi un « brin direct », ou « brin précoce », (leading strand) et un « brin indirect », ou « retardé », ou « tardif », (lagging strand) :
- le « brin direct » est le brin complémentaire du brin parental orienté 3’ vers 5’ (le « brin direct » est donc orienté 5’ vers 3’). Il est donc créé de façon continue, dans le sens 5’ vers 3’ ;
- le « brin indirect » est le brin complémentaire du brin parental orienté 5' vers 3’ (le « brin indirect » est donc orienté 3’ vers 5’). Il est créé de façon discontinue, sous forme de fragments d’Okazaki, dans le sens 5’ vers 3’.
-> les fragments d'Okazaki chez le procaryote mesure 1000 à 2000 bases, et chez l'eucaryote 200 bases
L'ADN polymérase a besoin d'une amorce pour fonctionner, parce qu'elle ne commence à synthétiser que par une extrémité 3'OH Libre. Et c'est l'amorce ARN qui va fournir cette extrémité libre. La présence ici d'ARN est expliquée par le fait que seules deux enzymes peuvent synthétiser les nucléotides : L'ARN Polymérase et L'ADN Polymérase. Dans le cas présent, l'ADN Polymérase ne pouvant fonctionner sans amorce, c'est L'ARN qui prend le relai pour fournir l'amorce nécessaire. La primase va en effet créer ces amorces d'ARN. Il y aura donc sur le brin retardé des jonctions ARN-ADN, qui seront par la suite éliminées par une RNase H. Des ADN polymérases particulières vont ensuite combler les lacunes laissées par l'ARN.
D'autres enzymes sont nécessaires au bon fonctionnement de la réplication. Les polymérases sont retenues à l'ADN parental par des protéines tenons et des protéines à pince coulissante. C’est le PCNA des eucaryotes et des archées, et la sous unité b des bactéries. Une hélicase brise les liaisons H entre les deux brins, et des protéines SSB se fixent à l'ADN monocaténaire par des liaisons salines, pour éviter la reformation de liaisons entre les deux brins. Sur l'ADN double brin précédant l'hélicase se fixe une topoisomérase I qui va permettre d'éviter les torsions entraînées par l'ouverture de la double-chaîne par l'hélicase (comme pour une ficelle dont on écarte les deux brins), en coupant un des brins, puis le ressoudant après déroulement. Une topoisomérase II va se fixer sur une des molécules d'ADN filles, et par la scission des deux brins de celle-ci, va permettre le démêlement des 2 ADN filles. Elle ressoude ensuite (après le passage de l'autre molécule dans l'interstice formé) la molécule lysée.vvvv
La terminaison
Cette phase correspond à l’arrêt de la réplication lorsque deux fourches de réplication se rencontrent ou lorsqu’une fourche rencontre un signal de terminaison de la réplication. Il y a "ter" : terA terD terB terC qui freine les fourches de réplication.