Séquençage de l'ADN - Définition
La liste des auteurs de cet article est disponible ici.
Introduction
Le séquençage de l'ADN consiste à déterminer l'ordre d'enchaînement des nucléotides d’un fragment d’ADN donné. Actuellement, la plupart des séquençages d’ADN sont réalisés par la méthode de Sanger décrite ci-dessous. Cette technique utilise la réaction de polymérisation de l’ADN à l'aide d'une ADN polymérase et des didésoxyribonucléotides (ddNTP).
La séquence d’ADN contient l’information nécessaire aux êtres vivants pour survivre et se reproduire. Déterminer cette séquence est donc utile aussi bien pour les recherches visant à savoir comment vivent les organismes que pour des sujets appliqués. En médecine, elle peut être utilisée pour identifier, diagnostiquer et potentiellement trouver des traitements à des maladies génétiques. En biologie l'étude des séquences d'ADN est devenue un outil important pour la classification des espèces.
Historique
Le séquençage de l'ADN a été inventé dans la deuxième moitié des années 1970. Deux méthodes ont été développées indépendamment, l'une par l'équipe de Walter Gilbert, aux États-Unis, et l'autre par celle de Frederick Sanger, en Grande-Bretagne. Ces deux méthodes sont fondées sur des principes diamétralement opposés : l'approche de Sanger est une méthode par synthèse enzymatique sélective, tandis que celle de Maxam et Gilbert est une méthode par dégradation chimique sélective. Pour cette découverte, Gilbert et Sanger ont été récompensés par le prix Nobel de chimie en 1980.
Initialement, la méthode de Sanger nécessitait de disposer d'un ADN simple brin qui servait de matrice pour la synthèse enzymatique du brin complémentaire. Pour cette raison, le premier organisme biologique dont le génome a été séquencé en 1977 est le virus bactériophage φX174. Ce virus a la propriété d'avoir un génome constitué d'ADN simple brin qui est encapsulé dans la particule virale.
Au cours des 25 dernières années, la méthode de Sanger a été largement développée grâce à plusieurs avancées technologiques importantes :
- La mise au point de vecteurs de séquençage adaptés, comme le phage M13 développé par Joachim Messing au début des années 1980.
- Le développement de la synthèse chimique automatisée des oligonucléotides qui sont utilisés comme amorces dans la synthèse.
- L'introduction de traceurs fluorescents à la place des marqueurs radioactifs utilisés initialement. Ce progrès à permis de sortir le séquençage des pièces confinées nécessaires à l'usage de radioisotopes.
- L'adaptation de la technique PCR pour le séquençage.
- L'utilisation de séquenceurs automatiques de gènes
- L'utilisation de l'électrophorèse capillaire pour la séparation et l'analyse
La méthode de Maxam et Gilbert nécessite des réactifs chimiques toxiques et reste limitée quant à la taille des fragments d'ADN qu'elle permet d'analyser (<250 nucléotides). Moins facile à robotiser, son usage est devenu aujourd'hui confidentiel.
Méthode de Maxam et Gilbert
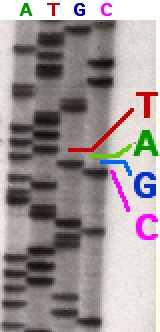
Cette méthode est basée sur une dégradation chimique de l'ADN et utilise les réactivités différentes des quatre bases A, T, G et C, pour réaliser des coupures sélectives. En reconstituant l'ordre des coupures, on peut remonter à la séquence des nucléotides de l'ADN correspondant. On peut décomposer ce séquençage chimique en six étapes successives :
- Marquage : Les extrémités des deux brins d'ADN à séquencer sont marquées par un traceur radioactif (32P). Cette réaction se fait en général au moyen d'ATP radioactif et de polynucléotide kinase
- Isolement du fragment d'ADN à séquencer. Celui-ci est séparé au moyen d'une électrophorèse sur un gel de polyacrylamide. Le fragment d'ADN est découpé du gel et récupéré par diffusion
- Séparation de brins. Les deux brins de chaque fragment d'ADN sont séparés par dénaturation thermique, puis purifiés par une nouvelle électrophorèse.
- Modifications chimiques spécifiques. Les ADN simple-brin sont soumis à des réactions chimiques spécifiques des différents types de base. Walter Gilbert a mis au point plusieurs types de réactions spécifiques, effectuées en parallèle sur une fraction de chaque brin d'ADN marqué. Par exemple une pour les G (alkylation par le diméthyle sulfate), une pour G et les A (dépurination), une pour les C, ainsi qu'une pour les C et les T (hydrolyse alcaline). Ces différentes réactions sont effectuées dans des conditions très ménagées, de sorte qu'en moyenne chaque molécule d'ADN ne porte que zéro ou une modification.
- Coupure. Après ces réactions, l'ADN est clivé au niveau de la modification par réaction avec une base, la pipéridine.
- Analyse. Pour chaque fragment, les produits des différentes réactions sont séparés par électrophorèse en conditions dénaturantes et analysés pour reconstituer la séquence de l'ADN. Cette analyse est analogue à celle que l'on effectue pour la méthode de Sanger.