Réaction en chaîne par polymérase - Définition
La liste des auteurs de cet article est disponible ici.
Introduction
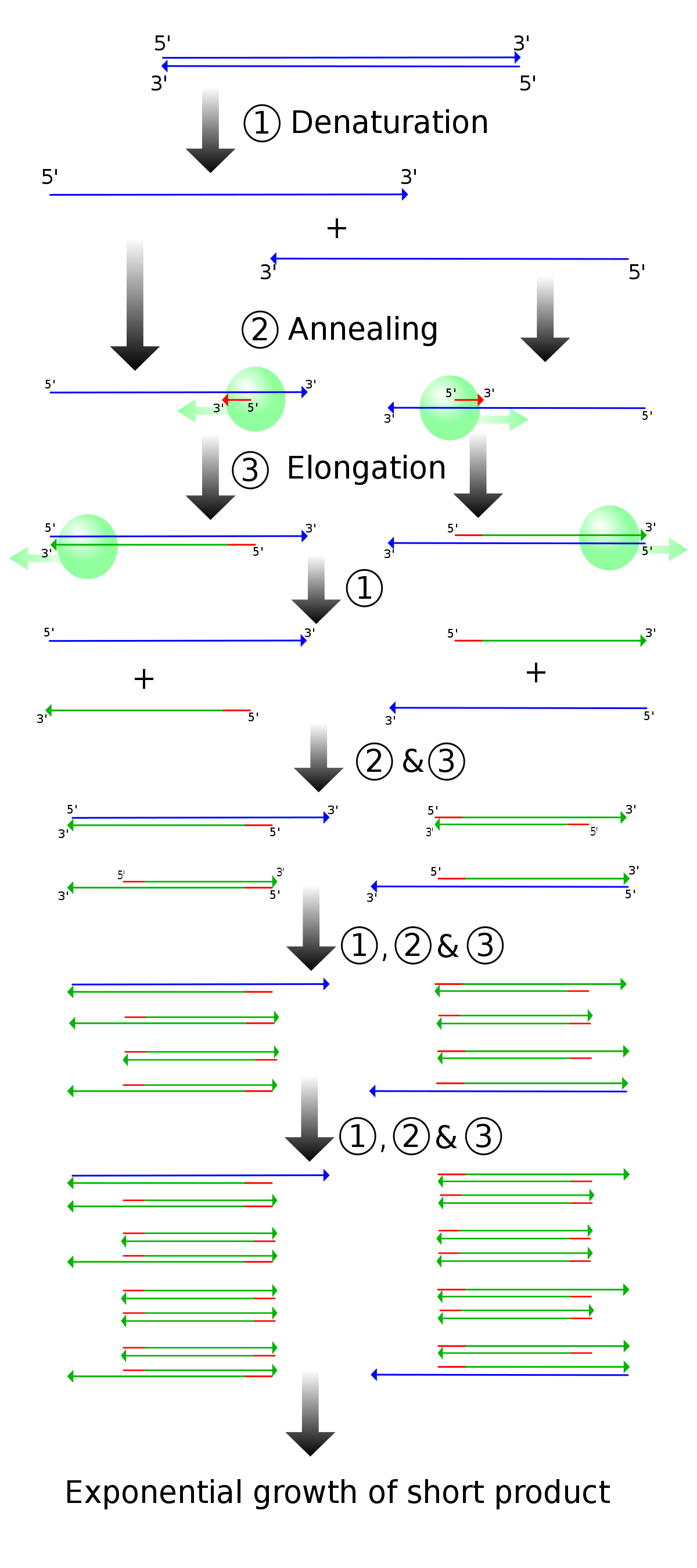
L'amplification en chaîne par polymérase ou réaction en chaîne par polymérase (PCR est l'abréviation anglaise de polymerase chain reaction, l'acronyme français ACP pour Amplification en Chaîne par Polymérisation est très rarement employé), est une méthode de biologie moléculaire d'amplification génique in vitro, qui permet de copier en grand nombre (avec un facteur de multiplication de l'ordre du milliard), une séquence d'ADN ou d'ARN connue, à partir d'une faible quantité (de l'ordre de quelques picogrammes) d'acide nucléique (séquence spécifique d’ADN (l’Amplicon) ou amorces spécifiques constituées d'oligonucléotides de synthèse de 20 à 25 nucléotides). Cette technique permet entre autres de détecter la présence du virus VIH ou de mesurer la charge virale (concentration du virus dans le plasma), des OGM (organismes génétiquement modifiés), des virus des hépatites B, C et D. De plus en plus utilisée en criminalistique, cette technique est basée sur la combinaison de deux facteurs :
- Les propriétés de synthèse enzymatique et d’initiation spécifique à l'ADN double brins spécifique des ADN polymérases dépendantes à l'ADN thermostables.
- Les propriétés d’hybridation et de déshybridation des brins complémentaires d’ADN en fonction de la température.
Ces éléments permettent de contrôler l’activité enzymatique grâce à des transitions de température (assurées par un thermocycleur) répétées de manière cyclique (cf. réaction en chaîne).
Les premières ADN polymérases utilisées provenaient d'une bactérie thermophile (résistante à des températures très élevées), par exemple Thermus aquaticus (Taq polymérase) ou encore Pyrococcus furiosus (Pfu polymérase), Thermococcus litoralis (Vent ou Tli polymérase), Thermus thermophilus (Tth polymérase). De nos jours, les enzymes utilisées sont dites recombinantes, ce qui simplifie considérablement leur obtention, et leurs propriétés ont été largement modifiées pour les rendre plus efficaces, plus fidèles…
En moins de dix ans, cette technique (maintenant capable de faire plus d'un milliard de copies en moins d'une heure) s'est imposée dans les laboratoires et a révolutionné la biologie moléculaire.
Historique
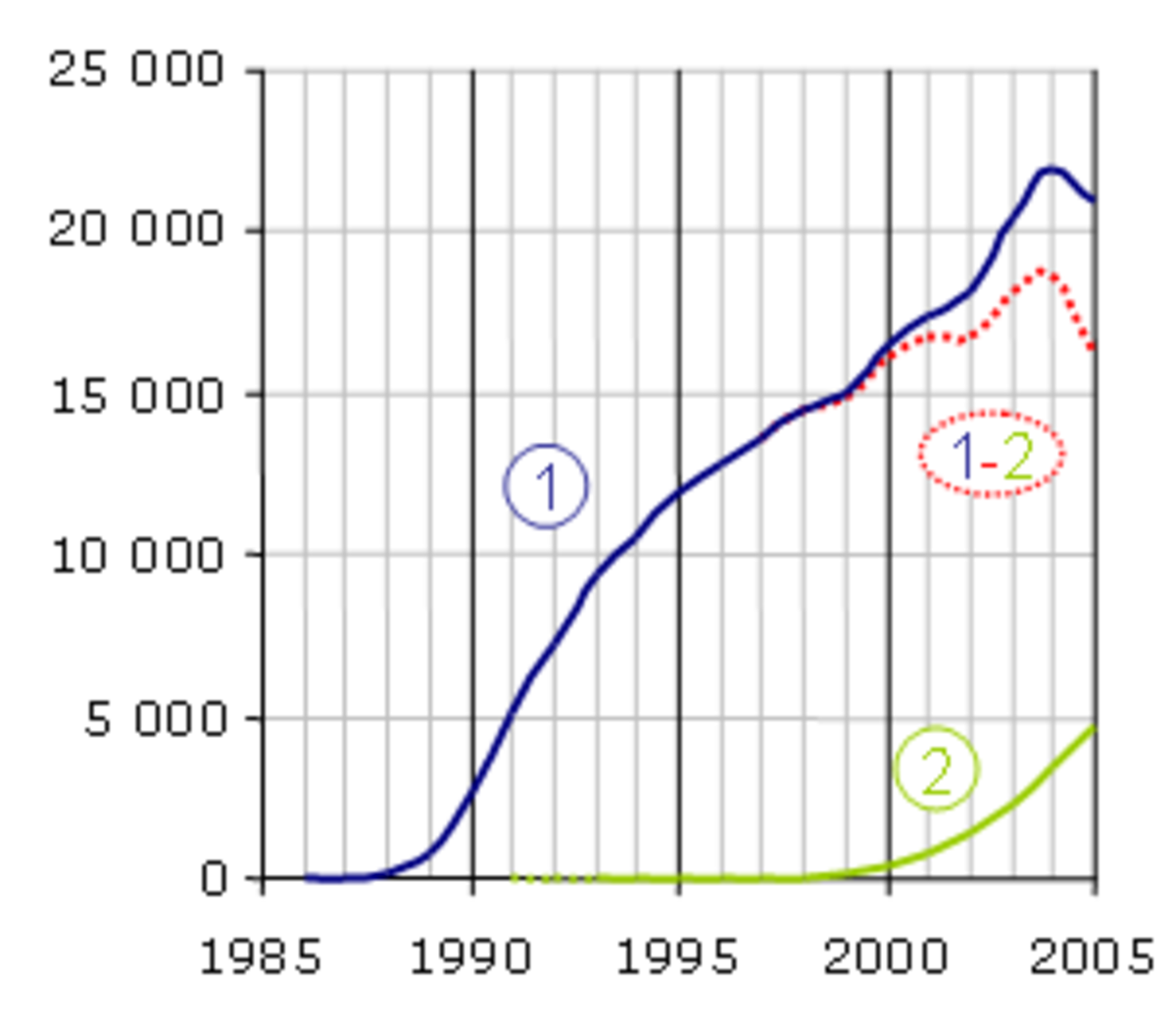
La PCR est une technologie qui a bouleversé la biologie moléculaire et s'est implantée très rapidement dans les laboratoires. En revanche, la PCR en temps réel a dû attendre la mise sur le marché d'un certain nombre d'innovations technologiques avant de se développer et est encore considérée comme une méthodologie nouvelle. Le nombre d'articles par année répondant aux mots clés (1) "polymerase chain reaction" et (2) "real-time polymerase chain reaction" sur le moteur de recherche PubMed donne une assez bonne idée de leur importance dans le monde scientifique. Notez que la méthode n'est pas exempte de biais, par exemple quelques articles sont trouvés pour la PCR en temps réel en 1991 et 1992 (en pointillé) alors que son principe n'a été décrit qu'en 1993. La différence (1-2) est représentative du poids de la PCR en point final, qui va probablement céder progressivement la place au temps réel.
Cette technique a largement évolué depuis ses débuts. Parmi les évolutions les plus fondamentales, on retrouve :
- Le remplacement des fragments de Klenow d’ADN polymérase I d’E . coli par une polymérase thermorésistante (initialement la Taq) qui évite de devoir remettre de l’enzyme à chaque cycle. Cette innovation permet un bond énorme vers l’automatisation et évite de devoir ouvrir le tube réactionnel, limitant considérablement le risque de contamination.
- La généralisation des thermocycleurs (un bon nombre d’anciennes expérimentations ont été réalisées avec trois bains-marie) a permis de rendre la PCR moins contraignante, plus reproductible et était un pré requis indispensable à la plupart des applications nouvelles.
- L’invention de la PCR en temps réel qui permet de rendre la méthode quantitative et évite plusieurs étapes expérimentales contraignantes, telles l’électrophorèse sur gel d’agarose, l’acquisition de fluorescence, la calibration de l’acquisition du signal, etc.
Par souci de clarté, les dates correspondent à la première publication sur le domaine et seuls les premiers auteurs sont cités, les références complètes étant dans le chapitre "bibliographie". Ce choix permet en outre de limiter les polémiques telles le rôle de Rosalind Elsie Franklin dans la découverte de la structure de la double hélice d’ADN.
- 1953 : Découverte de la structure en double hélice de l’ADN par James Dewey Watson et Francis Harry Compton Crick, (prix Nobel de physiologie ou médecine en 1962).
- 1956 : Découverte de l’ADN polymérase ADN dépendante (ADN pol I) par Arthur Kornberg (prix Nobel de physiologie ou médecine en 1959).
- 1970 : Co-découverte indépendante de l’ADN polymérase ARN dépendante par Temin HM et Baltimore D (prix Nobel de physiologie ou médecine en 1975).
- 1986 : Première publication publique sur la PCR par Kary Mullis (prix Nobel de chimie en 1993).
- 1988 : Première PCR réalisée avec une ADN polymérase thermostable, provenant de Thermus aquaticus, par Saiki RK.
- 1991 : Première détection du produit de PCR par sonde (sonde d’hydrolyse) par Holland PM.
- 1992 : Invention de la PCR en temps réel par Higuchi R.
- 1995 : Première publication sur la TAIL-PCR par Liu YG
- 1996 : Mise au point des polymérases temporairement inactives et activables par la chaleur par Birch DE.
- 1997 : Mise en évidence de la variation de température "Tm dépendante" du SYBR green par Wittwer CT.
- 1997 : Première discrimination d’allèle grâce à la courbe de fusion par Lay MJ.