Recombinaison génétique - Définition
La liste des auteurs de cet article est disponible ici.
La recombinaison au niveau moléculaire
Celle-ci nécessite la présence de séquences homologues entre deux régions de l'ADN.
- Modèle de Holliday
Le complexe RecBCD déroule l'ADN et coupe un brin à proximité d'un site chi. Une extrémité simple brin est produite et va envahir la molécule homologue. La protéine RecA va alors se charger de rechercher une séquence homologue : elle se fixe sur le simple brin avec SSB et réunit les deux séquences. La formation d'un hétéroduplex se termine par l'action de la ligase. Il se produit ensuite une rotation de l'hétéroduplex conduisant à la formation de la jonction de Holliday. Sous l'action de la protéine RuvAB, cette jonction va migrer et va être coupée (résolution) grâce à RuvC. Il y a deux coupures possibles conduisant soit à un enjambement (recombinaison entre allèles), soit à une zone hétéroduplex (la probabilité de survenue est identique).
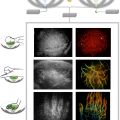
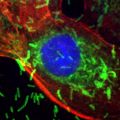
Complexe Rad51 : rôle de la protéine RecA.
La recombinaison homologue
Le terme recombinaison homologue, correspond à un évènement de recombinaison génétique entre deux séquences identiques situées sur 2 molécules d'ADN différentes, ou distantes l'une de l'autre sur la même molécule. Ce processus est très fréquent chez la levure et donc largement utilisé comme outil de biologie moléculaire dans ce cas.
La recombinaison illégitime
La recombinaison site-spécifique
-Etape de reconnaissance guidé par une enzyme de recombinaison. -L'appariement entre les deux molécules en cours de recombinaison n'est pas épigé. -L'enzyme de recombinaison reconnait des séquences nucléotidiques spécifiques.