Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
La fréquence accrue de tuberculose résistante aux antibiotiques met en péril le programme mondial de lutte contre la maladie. Une étude réalisée par un consortium international sous l'égide de l'Université d'Oxford et impliquant l'équipe de Philip Supply au Centre d'infection et d'immunité de Lille, démontre le potentiel du séquençage de génomes de l'agent infectieux comme nouvelle approche diagnostique, plus rapide et plus prédictive que les tests existants. Elle permettra une mise en place précoce de traitements mieux à même de guérir les cas de résistance. Ces travaux sont publiés dans la revue Lancet Infectious Diseases.
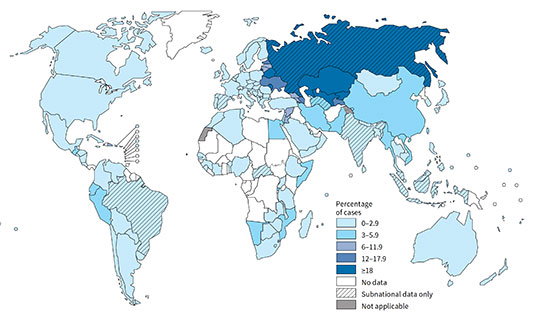
Figure: Distribution mondiale des proportions de nouveaux cas de tuberculose, contractée sous une forme multi-résistante aux antibiotiques. Les pourcentages indiqués se rapportent aux nombres totaux de nouveaux cas de tuberculose de toutes formes, basés sur l'année la plus récente (variable selon les pays) pour lesquels les données ont été rapportées.
© Organisation Mondiale de la Santé
A l'origine de près de 9 millions de nouveaux cas de maladie et 1.5 million de morts par an, la tuberculose demeure un problème majeur de santé publique. L'objectif de l'Organisation Mondiale de la Santé de stopper l'épidémie de tuberculose d'ici aux deux prochaines décennies, est menacé par le grand nombre de cas de formes multi- et ultra-résistantes aux traitements antituberculeux, estimés respectivement à près de 500000 et 50000 pour la seule année 2013. Les tests classiques par culture de l'agent bactérien Mycobacterium tuberculosis demeurent le standard pour tester les résistances. Cependant, ces tests peuvent prendre de nombreuses semaines et sont chers. Bien que plus rapides, les tests actuels d'analyse génétique ne couvrent qu'une fraction limitée des mutations bactériennes communément associées à certains types de résistance, et n'identifient ou n'excluent pas d'autres mécanismes de résistance.
Tirant parti des progrès impressionnants des techniques de séquençage d'ADN de nouvelle génération, l'étude du consortium international évalue la capacité d'une analyse moléculaire étendue à l'ensemble du génome de la bactérie à prédire la résistance ou la susceptibilité à 11 antibiotiques utilisés dans les traitements de première et seconde intention. Plus de 3600 génomes de souches bactériennes résistantes ou sensibles aux antibiotiques ont été séquencés, dont plus de 900 sur la base d'une collaboration de l'équipe de Philip Supply avec Genoscreen, une société implantée sur le campus de l'Institut Pasteur de Lille. En appliquant un algorithme de tri sur un groupe de 23 gènes connus pour être associés à différents type des résistance, la présence de mutations déterminant ou ne déterminant pas la résistance, ou de mutations non caractérisées, a été établie dans un premier sous-ensemble test de génomes. L'ensemble de 120 mutations déterminantes et de 772 mutations non déterminantes de résistance identifiées a ensuite servi pour évaluer la capacité à prédire la résistance ou la susceptibilité aux antibiotiques sur un second sous-ensemble de génomes, en comparaison des données phénotypiques établies sur la base des tests de culture standards des souches correspondantes. Près de 90% des profils phénotypiques ont pu être prédits sur cet ensemble, avec une précision supérieure à 90 %. Ces performances se sont avérées supérieures à celles combinées pour les trois tests moléculaires existants, interrogeant des panels beaucoup plus restreints de mutations.
Le large catalogue de mutations construit au travers de cette étude - destiné à s'étendre encore - permet donc d'utiliser cliniquement les données dérivées du séquençage génomique pour prédire la résistance ou la susceptibilité aux antibiotiques, bien plus rapidement que les tests de cultures standards et plus précisément que les tests génétiques actuels. Ces résultats, couplés au développement annoncé de nouvelles plates-formes de séquençage ultra-portables et peu onéreuses, pourraient transformer le diagnostic de résistance anti-tuberculeuse, y compris dans les pays à faibles revenus, où beaucoup de patients ne bénéficient que de traitements établis sur une base empirique, réduisant les chances de succès et augmentant les risques de développer des résistances additionnelles.
Populaires