Empreinte génétique - Définition
La liste des auteurs de cet article est disponible ici.
Principes généraux
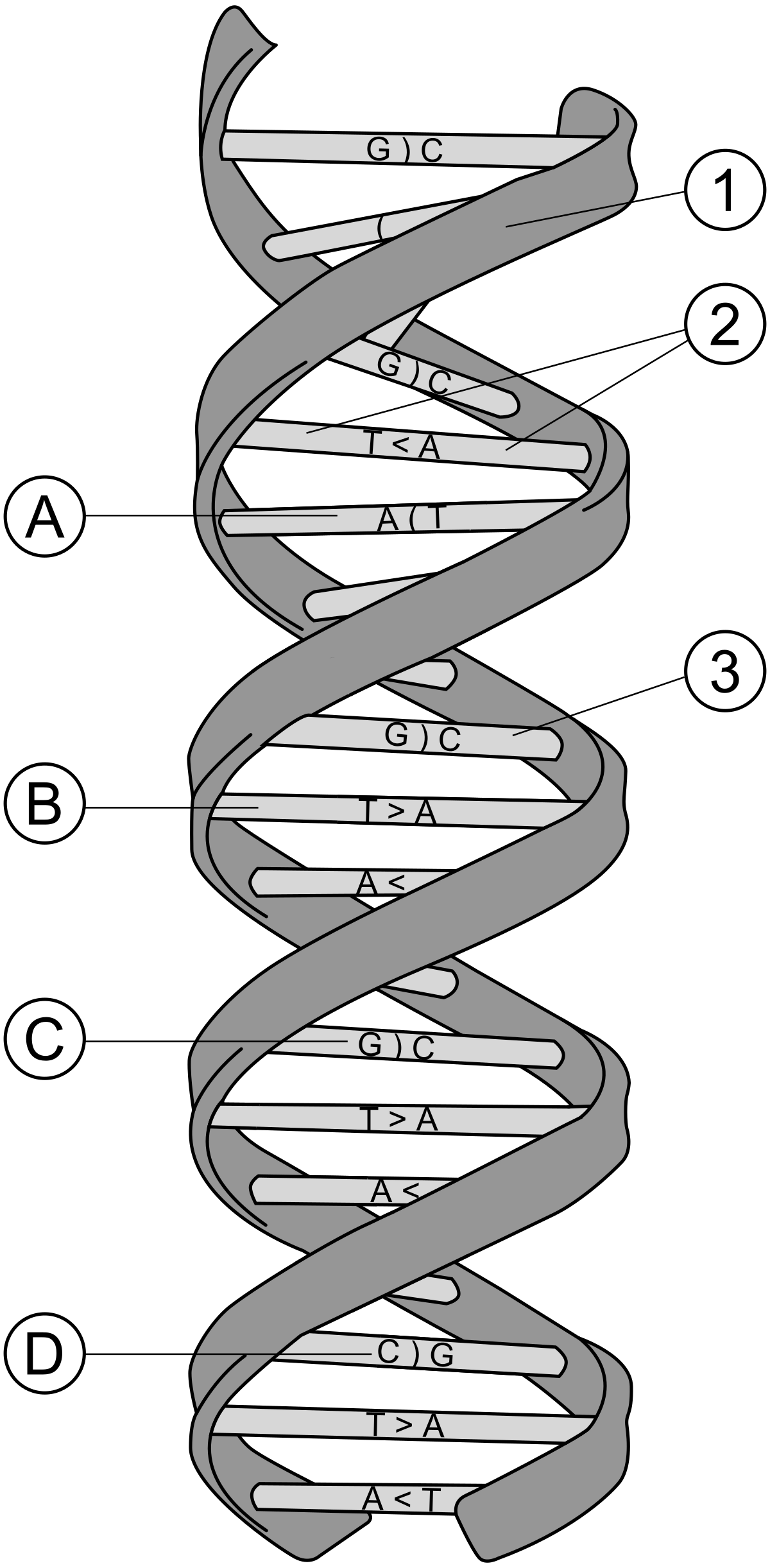
L'ADN est constitué de séquences de nucléotides parmi les quatre suivants : A (adénine), C (cytosine), T (thymine), G (guanine).
Il faut rappeler que les gènes permettent la fabrication des protéines. Mais il existe sur l'ADN des portions qui ne codent aucune protéine. Ce sont certaines d'entre elles, appelées microsatellites et minisatellites, qui sont spécifiques à chaque individu et constituent sa signature génétique.
Ce sont des séquences d'ADN répétées. Il y a :
- les séquences répétées en tandem courtes, appelées aussi microsatellites ou (STR) pour Short Tandem Repeats en anglais. La plupart des séquences répétées sont des répétions de 4 nucléotides, mais les longueurs de 2 à 5 bases sont aussi étudiés. Exemple :
| CTGG CTGG CTGG CTGG CTGG CTGG |
- les minisatellites ou VNTR pour « Variable Number Tandem Repeats » . Les séquences répétées sont des répétions de 10 à 100 nucléotides. On regroupe parfois ces méthodes sous le nom « Multiple Loci VNTR Analysis » (Mlva).
Ces régions de l’ADN sont très polymorphes : en effet, le nombre de répétitions est variable pour chaque individu. Parce que les gens n’ont pas le même nombre de répétions, ces régions de l’ADN permettent d'identifier les individus.
La région des chromosomes où se situent ces séquences ( leurs locus) doivent être repérées, puis amplifiés par PCR: il s'agit de fabriquer de nombreuses copies de cet ADN pour que celui-ci soit « visible » à la fin de l'analyse. Les fragments d'ADN obtenus sont séparés et identifiés par électrophorèse. Ceci permet de connaître leur longueur et donc d'en déduire le nombre de répétitions.
Les 2 méthodes de séparations les plus communes sont l’électrophorèse capillaire et électrophorèse sur gel.
Fiabilité
La grande force des systèmes basés sur les microsatellites est la fiabilité statistique de l’identification.
Le polymorphisme de chaque microsatellite est par lui-même très variable. Une version d'un même locus (ou allèle) peut avoir une fréquence comprise entre 5 et 20 % des individus. Un seul locus ne permet donc pas de désigner un individu précis . Il faut utiliser plusieurs loci.
En France et aux États-unis, on utilise couramment 13 loci (régions de séquence répétée) pour une identification. Et comme chaque locus est composé d’une certaine séquence de répétition (microsatellite) et que le nombre de répétition est parfaitement indépendant des répétitions sur les autres loci, les règles de statistiques peuvent être appliqués.
Par exemple, pour trois loci A, B et C, indépendants, et pour lesquels il existe plusieurs versions ( A1,A2,A3, et B1, B2,...) , on peut dire que Probabilité (A1,B2, C1)=Probabilité (A1) x Probabilité (B2,) x Probabilité (c1). Ainsi pour 13 loci, la probabilité d’avoir deux séquences identiques pour 2 individus différents non apparentés est estimée à 1 chance sur 1018 , ce qui est quasiment négligeable (très proche de zéro). Par conséquent, plus le nombre de microsatellites analysés est important, plus l’identification est fiable. Toutefois, dans le cas général, si on ne sait pas si les 2 individus sont apparentés, la probabilité monte à 1 chance sur 3x1012 (parce que 0,2% de la population mondiale est constituée de jumeaux monozygotes).
Selon les pays différents, différents systèmes d’identification ADN base sur la répétition des microsatellites sont utilisés. En Amérique du Nord (USA, Canada), la norme CODIS est la plus utilisée, alors qu’en Angleterre, c’est le SGM+.En France, les empreintes génétiques sont rassemblées dans le Fichier national automatisé des empreintes génétiques (FNAEG), destiné à l'origine à recueillir les empreintes des personnes condamnées pour infractions à caractère sexuel, mais dont l'usage s'est rapidement étendu à toutes sortes de délit, contenant en 2008 plus de 700 000 profils (soit près de 1% de la population française).
Mais de toute manière, plusieurs zones de microsatellites sont communes aux différentes normes utilisées, ce qui permet la compatibilité entre elles.