Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
En raison de la petite taille de leur génome, les virus de plantes multiplient les stratégies d'expression pour produire les protéines nécessaires à leur prolifération. L'équipe de Véronique Ziegler-Graff à l'Institut de biologie moléculaire des plantes, en collaboration avec une équipe de bioinformaticiens de l'université de Cambridge (Grande-Bretagne) et un chercheur de l'Iowa State University, Ames (USA) a découvert chez un virus de la famille des Luteoviridae, un nouveau gène de petite taille codant une protéine indispensable au transport viral dans la plante et resté inaperçu jusqu'à présent en raison de son mode non conventionnel de traduction. Ces travaux sont publiés dans la revue PLoS Pathogens.
Les Luteoviridae sont des virus qui infectent de nombreuses plantes d'intérêt agronomique comme les céréales, la pomme de terre, la betterave à sucre ou le colza. Ces maladies virales occasionnent d'importantes pertes économiques et des traitements insecticides sont déployés pour réduire l'impact de ces virus transmis par des pucerons. Pour se soustraire aux effets néfastes de ces traitements sur l'homme et l'environnement, il est urgent de développer des stratégies de lutte innovantes contre ces virus. L'étude approfondie de leur cycle d'infection devrait permettre d'identifier de nouveaux angles d'attaque.
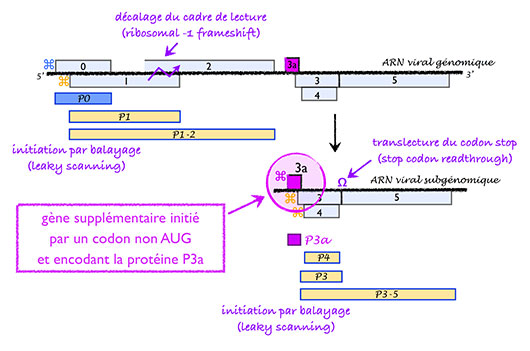
Figure: Organisation génomique d'un polerovirus (famille des Luteoviridae), illustrant la diversité des stratégies d'expression développées pour exprimer plusieurs protéines à partir d'un seul ARN viral: leaky scanning, ribosomal frameshift, synthèse d'un ARN subgénomique, translecture du codon stop. Les rectangles clairs annotés de 0 à 5 positionnés de part et d'autre de l'ARN viral (trait noir épais) représentent les gènes portés par ce virus. Les protéines encodées (identifiées P0 à P3-5) sont schématisées par des rectangles présents sous les ARN génomiques et subgénomiques. Les différentes stratégies de traduction ainsi que les codons d'initiation de la traduction et leur contexte, mis en oeuvre par le virus sont précisés selon les symboles ci-dessous.

Avec six gènes partiellement chevauchants, le génome à ARN des Luteoviridae est un exemple de compaction de l'information génétique (voir Figure). Initiation de la traduction par balayage (leaky scanning), décalage du cadre de lecture (frameshift), translecture d'un codon de terminaison (readthrough) sont quelques-uns des mécanismes déployés par les Luteoviridae pour augmenter leur capacité d'encodage. La moitié 3' du génome est exprimée via un ARN subgénomique (ARN colinéaire en 3' à l'ARN génomique) qui encode trois protéines impliquées dans l'encapsidation, le mouvement du virus dans la plante et la reconnaissance spécifique des particules virales lors de la transmission par puceron. C'est au niveau de l'extrémité 5' de cet ARN subgénomique qu'une analyse bioinformatique a permis d'identifier une petite région présentant une capacité codante très conservée parmi l'ensemble des membres de la famille des Luteoviridae. Etonnamment, ce gène permet la synthèse d'une petite protéine (5kDa) appelée P3a, bien qu'il ne possède pas de codon d'initiation de la traduction universel, à savoir un codon AUG, mais un codon alternatif de type ACG, AUU, AUA ou CUG, selon les virus.
L'équipe de Véronique Ziegler-Graff de l'Institut de biologie moléculaire des plantes a mis en oeuvre une approche de génétique inverse pour étudier le rôle de la protéine P3a du virus de la jaunisse du navet (Turnip yellows virus, genre polerovirus). La protéine P3a a été détectée grâce à des anticorps spécifiques dans les feuilles inoculées avec le virus, confirmant la fonctionnalité du codon d'initiation non conventionnel. Les chercheurs ont démontré l'importance de la protéine pour le mouvement systémique du virus dans l'ensemble de la plante en empêchant l'expression du gène 3a par mutagénèse dirigée. En effet, le virus modifié conserve sa capacité à se multiplier au site d'inoculation, mais perd son potentiel à envahir la plante entière. Exprimée sous la forme d'une protéine de fusion avec le marqueur fluorescent GFP, la protéine a été détectée au niveau des plasmodesmes de la cellule (pores dans la paroi cellulaire permettant aux virus de passer d'une cellule à l'autre), renforçant ainsi l'hypothèse de son rôle dans la propagation du virus dans la plante.
Au-delà de l'identification d'un gène cryptique essentiel au cycle infectieux des virus de la famille des Luteoviridae, cette découverte laisse entrevoir l'existence chez d'autres virus, voire chez d'autres pathogènes, de gènes passés encore inaperçus, car traduits à partir de codons d'initiation non conventionnels
Populaires