Puce à ADN - Définition
La liste des auteurs de cet article est disponible ici.
Introduction
Une puce à ADN est un ensemble de molécules d'ADN fixées en rangées ordonnées sur une petite surface qui peut être du verre, du silicium ou du plastique. Cette biotechnologie récente permet d'analyser le niveau d'expression des gènes (transcrits) dans une cellule, un tissu, un organe, un organisme ou encore un mélange complexe, à un moment donné et dans un état donné par rapport à un échantillon de référence.
Les puces à ADN sont aussi appelées puces à gènes, biopuces, ou par les termes anglais « DNA chip, DNA-microarray, biochip ». Les termes français microréseau d'ADN et micromatrice d'ADN sont recommandés par l'Office québécois de la langue française.
Le principe de la puce à ADN repose sur la propriété que possède l'ADN dénaturé de reformer spontanément sa double hélice lorsqu'il est porté face à un brin complémentaire (réaction d'hybridation). Les quatre bases azotées de l'ADN (A, G, C, T) ont en effet la particularité de s'unir deux à deux par des liaisons hydrogènes (A = T et T = A ; G ≡ C et C ≡ G). Si un patient est porteur d'une maladie, les brins extraits de l'ARN d'un patient (et rétrotranscrits en ADN), vont s'hybrider avec les brins d'ADN synthétiques représentatifs de la maladie.
Principe
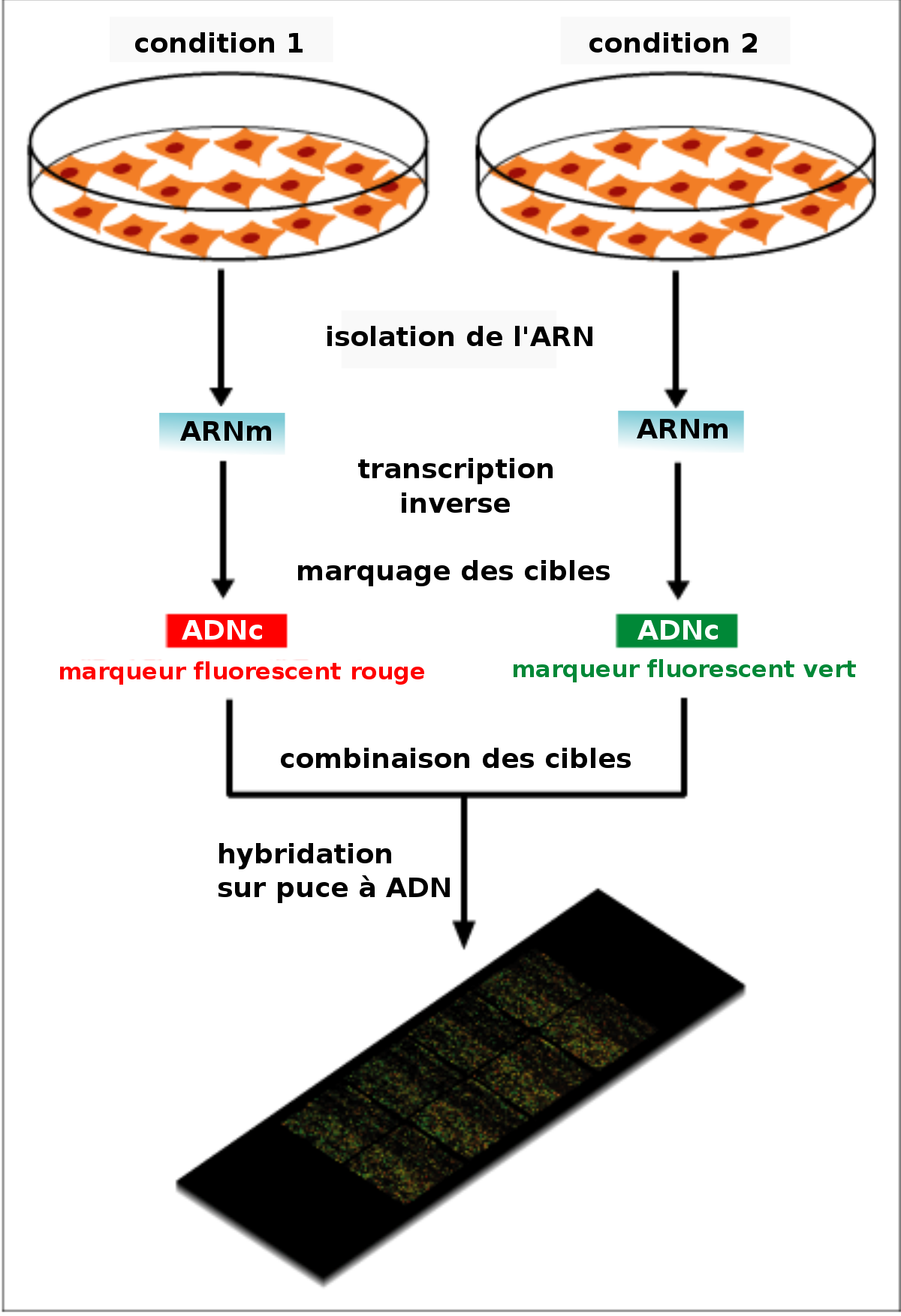
Concrètement, les ARN totaux sont extraits de cellules, dont on veut comparer l'expression des gènes avec un étalon, et subissent une amplification qui va permettre d'obtenir une quantité de matériel génétique suffisante pour l'expérience. Ensuite ces ARNm sont transformés en ADN complémentaires (ADNc) par la technique de rétrotranscription et marqués par un colorant (soit la Cyanine 3 (fluorochrome vert) soit la Cyanine 5 (fluorochrome rouge)). On met ensuite les ADNc obtenus dans une puce contenant des fragments d'ADN, en même temps que l'ADNc étalon. Chaque point (ou spot) de la puce va être analysé individuellement par un scanner à très haute résolution, et ce à la longueur d'onde d'excitation de la Cyanine 3 puis de la Cyanine 5. L'image scannée va être traduite en niveaux de gris. On va ensuite comparer l'intensité du signal entre le vert et le rouge. En fonction de l'intensité du signal il y aura plus ou moins de pixels pour chaque point de la puce. A chaque point (ou spot) est attribué une valeur d'intensité normalisée par rapport à l'ADN "étalon" : on parle de spike. Chacune des valeurs peut être analysée par des techniques de bio-informatique, ce qui permet d'estimer avec plus ou moins de précision l'intensité d'expression d'un gène. Selon les techniques de biologie moléculaire, un marquage à la biotine des ADNc est possible mais dans ce cas pour comparer deux populations ou deux tissus, il faudra hybrider pour chaque condition une puce (et non pas les deux marquages sur la même puce en compétition.)
Par exemple on peut marquer l'ADN complémentaire du malade en vert et du traité en rouge, ou bien, du témoin en rouge et du traité en vert. Ce marquage se fait habituellement grâce à une enzyme : la polymérase T7 qui amplifie l'ARNm et incorpore les cyanines pour un marquage optimal. Une fois marqués ces ADN complémentaires sont déposés sur la lame de verre qui, elle-même, possède fixée à sa surface, des fragments de génome humain recouvrant tous les gènes présents dans une cellule.
Les molécules d'ADN fixées sur la lame sont appelées des sondes même si la nomenclature peut varier. Des dizaines de milliers de sondes peuvent être fixées sur une même puce. Cela permet de tester différentes cultures cellulaires sur une même lame voire de faire des réplicats (ce qui est vivement recommandé pour l'analyse biostatistique en aval). Cette technologie provient d'une adaptation du Northern Blot où de l'ADN fragmenté est fixé à un support puis hybridé avec un ARNc. La mesure de l'expression de gènes par puce à ADN s'applique à de nombreux domaines de la biologie et de la médecine comme l'étude de traitements, de maladies ou bien encore de stades développementaux.