Acide ribonucléique - Définition
La liste des auteurs de cet article est disponible ici.
Fonction dans la cellule
Dans les cellules, les ARN remplissent quatre rôles distincts et complémentaires :
- Support temporaire de l'information génétique. C'est l'ARN messager qui remplit ce rôle, il est utilisé par la cellule pour transmettre l'information correspondant à un gène donné à l'extérieur du noyau, puis pour synthétiser des protéines à partir de ces informations ;
- Catalyseur enzymatique. Comme les protéines, les ARN peuvent se replier en trois dimensions pour former des structures complexes. Ces structures permettent à certains ARN de se comporter comme des enzymes, on parle alors de ribozyme. Le ribosome, la ribonucléase P et certains introns sont des ribozymes. Il existe des arguments indirects indiquant que la machinerie d'épissage des ARN messagers (le splicéosome) est également aussi un ribozyme, même si la démonstration formelle n'en a pas encore été apportée ;
- Guide pour des enzymes. Certains ARN sont utilisés comme cofacteurs par des protéines pour permettre leur ciblage vers des séquences spécifiques. Parmi ceux-ci, on peut citer les petits ARN nucléolaires (snoARN), qui guident les enzymes de modification de l'ARN ribosomique, l'ARN télomérique, qui est un cofacteur de la télomérase, l'enzyme qui fabrique les extrémités des chromosomes, ou encore les ARN interférents ;
- Régulateurs de l'expression génétique. Certains ARN non codants jouent un rôle dans la répression de l'expression de certains gènes ou groupes de gène. C'est le cas par exemple des ARN antisens qui s'apparient à un ARN cible et en bloquent la traduction par le ribosome.
Une classe particulière d'ARN, les ARN de transfert, se trouve à l'interface de plusieurs de ces fonctions en guidant les acides aminés lors de la traduction.
Enfin, le génome de certains virus est exclusivement constitué d'ARN et pas d'ADN. C'est en particulier le cas des virus de la grippe, du SIDA, de l'hépatite C et de la poliomyélite. Suivant les cas, la réplication de ces virus peut passer par un intermédiaire ADN (rétrovirus), mais peut aussi se faire directement d'ARN en ARN.
L'ARN est donc une molécule très polyvalente, ce qui a conduit Walter Gilbert, co-inventeur du séquençage de l'ADN à proposer en 1986 une hypothèse selon laquelle l'ARN serait la plus ancienne de toutes les macromolécules biologiques. Cette théorie, dite du « RNA World » (le monde de l'ARN), permet de s'affranchir d'un paradoxe de l'œuf et de la poule qui survient lorsqu'on cherche à savoir qui des protéines (catalyseurs) et de l'ADN (information génétique) sont apparus en premier. Dans ce modèle, l'ARN, capable de combiner à la fois les deux types de fonctions, serait le précurseur universel.
Les ARN messagers
L'information génétique contenue au sein de l'ADN n'est pas utilisée directement par la cellule pour fabriquer des protéines. Celle-ci utilise pour cela des copies transitoires de l'information génétique que sont les ARN messagers ou ARNm. Chaque ARN messager porte un ou, parfois, plusieurs cistrons, c'est-à-dire les instructions pour former une seule protéine. Il correspond donc à la copie d'un seul des gènes du génome (on parle alors d'ARNm monocistronique) ou parfois de quelques-uns (ARNm polycistronique).
L'ARN messager ne contient la copie que d'un seul des deux brins de l'ADN, celui qui est codant, et pas la séquence complémentaire. Par rapport à la séquence du gène contenue dans l'ADN du génome, celle de l'ARNm correspondant peut contenir des modifications et en particulier l'épissage (voir plus haut), qui élimine les régions non codantes. L'ARN messager synthétisé dans le noyau de la cellule est exporté dans le cytoplasme pour être traduit en protéine. Contrairement à l'ADN, qui est une molécule pérenne, présente pendant toute la vie de la cellule, les ARN messagers ont une durée de vie limitée, de quelques minutes à quelques heures, après quoi ils sont dégradés et recyclés.
Un ARN messager comporte trois régions distinctes : une région 5' non-traduite ou 5'-UTR, située en amont du ou des cistrons qu'il porte ; une région codante correspondant à ce ou à ces cistrons ; et enfin, une région 3' non-traduite.
La fonction des ARN messagers est multiple. Ils permettent d'une part de préserver la matrice d'ADN originale, qui n'est pas directement utilisée pour la traduction, la cellule ne travaillant que sur la copie d'ARNm. L'existence d'ARN messagers offre surtout à la cellule un mécanisme crucial de régulation du cycle de production des protéines à partir du génome. Le besoin cellulaire en telle ou telle protéine peut varier en fonction de l'environnement, du type de cellule, du stade de développement. La synthèse protéique doit donc être activée ou arrêtée en fonction des conditions cellulaires. La régulation de la transcription de l'ADN en l'ARNm répond à une nécessité et est contrôlée par des facteurs de transcription spécifiques agissant sur les promoteurs des gènes cibles. Lorsque la quantité d'une protéine donnée est suffisante, la transcription d'ARNm est inhibée, celui-ci est progressivement dégradé et la production protéique cesse. Il est donc important que l'ARNm soit une molécule transitoire, afin de pouvoir réaliser cette régulation essentielle.
Les ARN de transfert
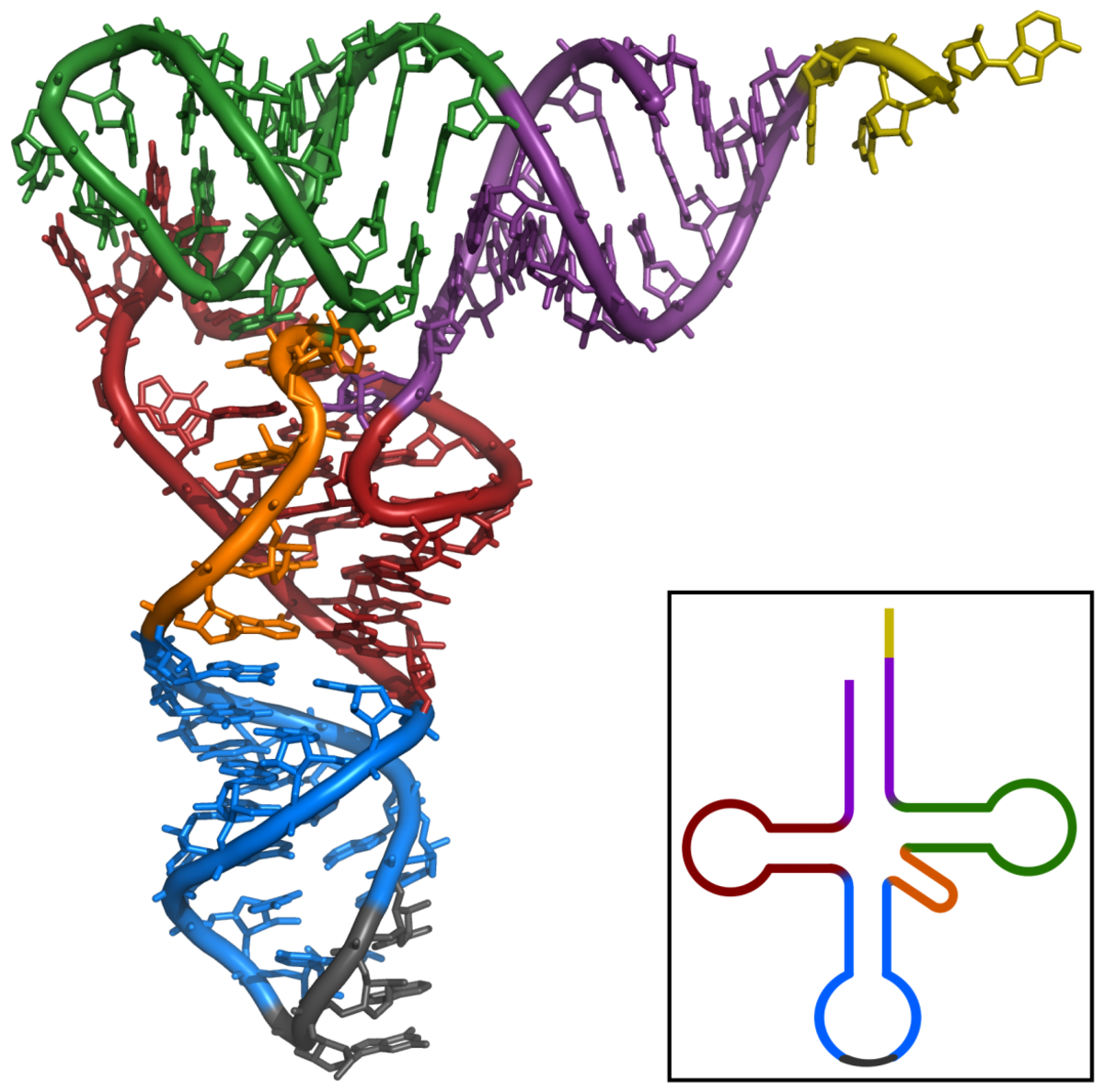
site de fixation de l'acide aminé en orange ;
branche D en rouge ;
boucle de l'anticodon en bleu, anticodon en noir ;
boucle T en vert.
Les ARN de transfert ou ARNt sont de courts ARN, longs d'environ 70 à 100 ribonucléotides, impliqués dans l'adressage des acides aminés vers les ribosomes lors de la traduction. Les ARN de transfert ont une structure caractéristique en feuille de trèfle, composée de quatre tiges appariées. L'une de ces tiges est terminée par une boucle qui contient l'anticodon, le triplet de nucléotides qui s'apparie au codon lors de la traduction d'un ARNm par le ribosome. À l'autre extrémité, l'ARNt porte l'acide aminé correspondant attaché par une liaison ester à son extrémité 3'-OH. Cette estérification est catalysée par des enzymes spécifiques, les aminoacyl-ARNt synthétases. En trois dimensions, la structure en feuille de trèfle se replie en « L », avec l'anticodon à une extrémité et l'acide aminé estérifié à l'autre extrémité.
Toutes les cellules vivantes contiennent un ensemble d'ARNt différents portant les différents acides aminés et capable de lire les différents codons.
Les ARN de transfert sont parfois désignés comme des « adapteurs » entre le code nucléique et le code protéique. C'est Francis Crick qui a proposé l'existence de ces adapteurs, avant même leur découverte en 1958.
Les ARN catalytiques ou ribozymes
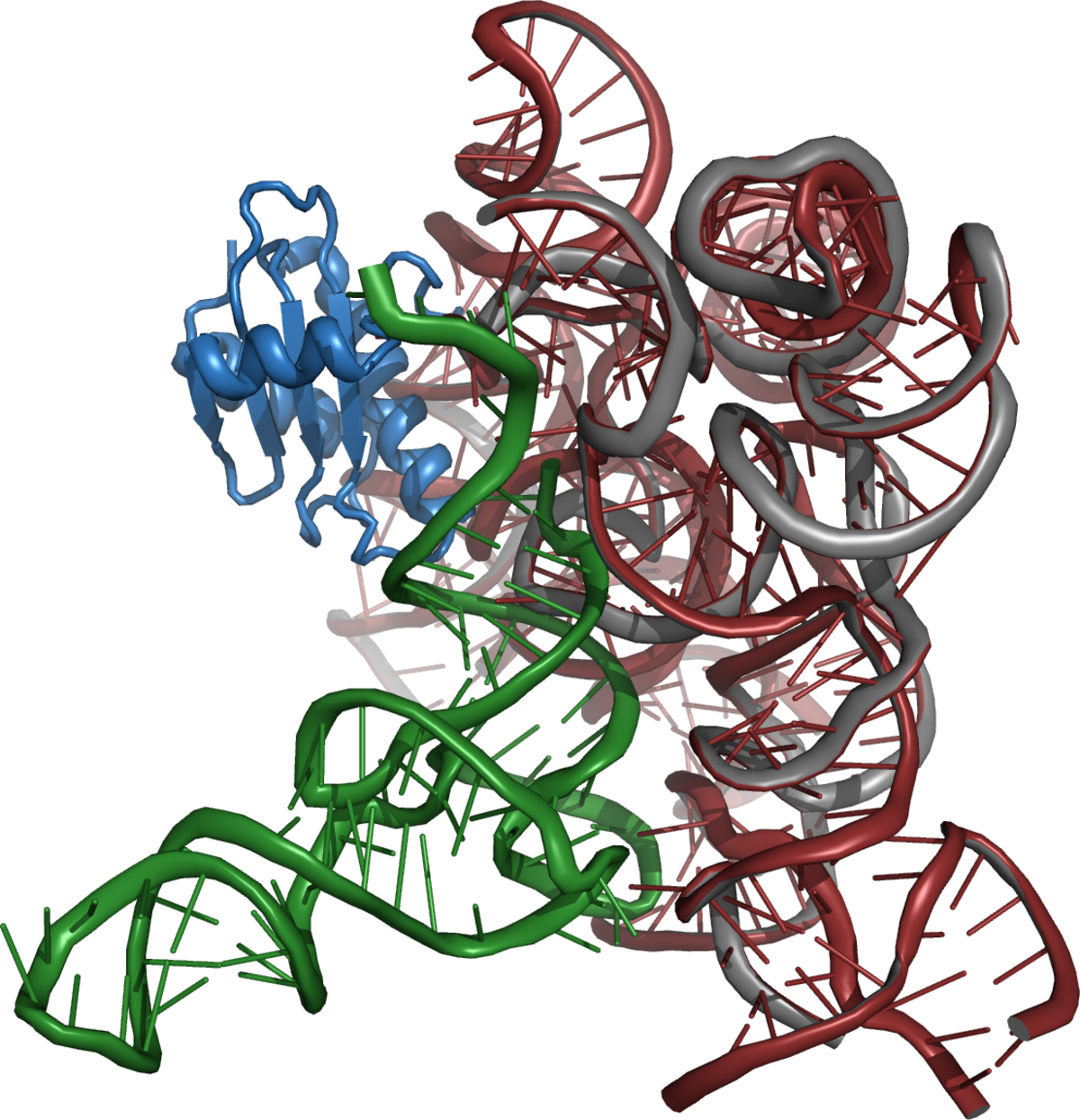
La découverte d'ARN possédant des capacités catalytiques a été faite dans les années 1980, en particulier par l'équipe de Thomas Cech, qui travaillait sur les introns du gène de l'ARN ribosomique du protozoaire cilié Tetrahymena, et celle de Sidney Altman, qui étudiait la ribonucléase P, l'enzyme de maturation de l'ARNt. Cech et Altman ont été récompensés par le prix Nobel de chimie en 1989 pour cette découverte.
Dans ces deux cas, l'ARN seul est capable de catalyser une réaction de coupure ou de transestérification spécifique, en l'absence de protéine. Ces ARN catalytiques ont été appelés ribozymes, car ce sont des enzymes constituées d'acide ribonucléique. Dans le cas de l'intron de Tetrahymena, il s'agit d'un auto-épissage, l'intron étant son propre substrat, tandis que la ribonucléase P est une enzyme agissant en trans, sur des substrats multiples.
Depuis ces découvertes initiales, d'autres ribozymes naturels ont été identifiés :
- les ARN de viroïdes ou de virus satellites (virusoïdes) qui sont capables de se cliver eux-mêmes ;
- il existe aujourd'hui des arguments très forts, basés sur la résolution de sa structure 3D, pour affirmer que ribosome, la machinerie cellulaire responsable de la traduction de l'ARNm en protéines, est lui-même un ribozyme. Les deux sites actifs du ribosome, le centre de décodage sur la petite sous-unité et le centre peptidyl-transférase qui forme les liaisons peptidiques, sont en effet exclusivement composés de segments d'ARN ribosomique ;
- le splicéosome, qui catalyse l'épissage des ARNm cytoplasmiques eucaryotes, est probablement aussi un ribozyme ;
- certains riboswitchs, des régions régulatrices structurées portées par des ARN messagers, ont une activité catalytique de coupure en présence d'un ligand ;
- il existe enfin des ribozymes synthétiques, qui ont été isolés par des méthodes d'évolution in vitro, comme la technique SELEX. On a ainsi pu isoler des ARN catalytiques synthétiques capables de catalyser une grande variété de réactions chimiques et de fixer des ligands très divers, ce qui a été interprété comme un argument en faveur de l'hypothèse du monde à ARN. De tels ARN synthétiques sont parfois appelés des aptamères, puisqu'ils sont « aptes » à réaliser une tâche donnée.
De manière générale, dans tous ces ribozymes, c'est leur repliement spécifique qui leur permet d'effecteur la reconnaissance de leur substrat et la catalyse, comme dans le cas des enzymes protéiques.
Les ARN guides
Les ARN guides sont des ARN qui s'associent à des enzymes protéiques et servent à en guider l'action sur des ARN ou des ADN de séquence complémentaire. L'ARN guide s'apparie à l'acide nucléique substrat et permet de cibler l'activité de l'enzyme. On a identifié plusieurs types :
- les petits ARN nucléolaires. On trouve, dans le nucléole des cellules eucaryotes, des ARN guides appelés petits ARN nucléolaires, ou snoARN, qui dirigent l'action d'enzymes de modification de l'ARN ribosomique, en particulier les 2'-O-méthylations par les snoARN C/D et les pseudouridylations par les snoARN H/ACA. Ce mécanisme permet à la cellule de modifier spécifiquement de multiples positions de l'ARNr, avec une seule enzyme en utilisant différents snoARN comme guides. Les snoARN sont souvent codés par des séquences introniques ;
- les microARN sont également des ARN guides qui interviennent dans le processus d'interférence ARN. Associé à un complexe protéique appelé RISC (RNA induced silencing complex), ces petits ARN provoquent soit une dégradation de l'ARNm cible auquel ils s'apparient, soit une répression de sa traduction ;
- TERC (« telomerase RNA component »), la sous-unité ARN de la télomérase. Cet ARN structuré est associé à la transcriptase inverse qui synthétise les télomères, extrémités des chromosomes. Il contient une séquence qui sert de substrat à la télomérase pour synthétiser l'ADN télomérique de séquence complémentaire. Il guide donc l'activité de l'enzyme, mais en servant de matrice, plutôt qu'en formant un appariement avec le substrat.
- chez les mammifères, les lincARN sont de larges ARN intergéniques, non codants, mais transcris comme les ARNm par l'ARN polymérase II. Leur longueur leur permet d'adopter une structure complexe tridimensionnelle. Ces structures complexes permettent leur intéraction avec différent cofacteurs transcriptionnels tel que hnRNP-K ou PRC2(principalement des inhibiteurs de la transcription). Ces complexes sont ensuite guidés grâce aux lincARNs sur les séquences régulatrices des gènes afin d'inhiber leur expression. La liaison des lincARN avec l'ADN impliquerait un appariement des bases ARN avec les bases ADN correspondante après désappariement de la double hélice d'ADN, voir la formation de triple hélice ADN-ADN-ARN. ;
Les ARN régulateurs

Certains ARN jouent un rôle de régulateurs directs de l'expression génétique. C'est en particulier le cas d'ARN non codants possédant des régions complémentaires d'ARN messagers cellulaires et qui peuvent donc s'y apparier pour former localement un duplex d'ARN. Ces ARN antisens peuvent être issus du même locus génétique que leur ARN cible, par transcription du brin complémentaire, on parle alors d'ARN cis-régulateurs. Ils peuvent aussi être issus de la transcription d'une autre région du génome, ce sont alors des ARN trans-régulateurs.
L'appariement de l'ARN régulateur avec son ARN messager cible peut agir sur la capacité de ce dernier à être traduit par le ribosome ou sur sa stabilité, ce qui aboutit à une régulation de la traduction du ou des gènes portés par l'ARN messager. Chez les bactéries, il existe ainsi de nombreux exemples d'ARN antisens cis- ou trans-régulateurs qui bloquent le site de démarrage de la traduction. Par exemple, le gène codant pour la porine OmpF est régulé par un ARN antisens appelé MicF.
Chez les eucaryotes, il existe aussi de grands ARN régulateurs, qui interviennent dans des processus de régulation épigénétique. L'exemple le mieux connu est celui de l'ARN Xist chez les mammifères. Celui-ci inactive non pas un gène, mais un chromosome entier. Xist recouvre l'un des deux chromosomes X de chaque cellule chez les individus femelles qui devient ainsi inactif. Un seul des deux chromosomes de la paire XX est ainsi actif, ce qui permet d'avoir le même taux d'expression des gènes portés par le chromosome X que chez les individus mâles, qui n'en ont qu'un. L'inactivation du X est un processus aléatoire, ce qui peut conduire à l'expression de différents phénotypes par différentes cellules, chez la même femelle. C'est par exemple le cas pour la couleur du pelage chez les chattes.